スクリーニング
この章では、Sievgeneを使ってスクリーニングを行う手順について解説します。
この章の内容
スクリーニングを行う前に
スクリーニング用の分子データを無料で配布しております。配布をご希望の場合は弊社までご連絡ください。
スクリーニング機能の利用には、計算サーバーに予めIBM Platform LSF/Torque/Slurmのいずれかをインストールする必要があります。また、弊社が配布しているスクリーニング用の分子データを計算サーバーの任意のディレクトリーに展開しておいてください。
▲ 上に戻る
計算サーバーへの接続
リボンの[スクリーニング]タブの[スクリーニング]ボタンをクリックします。
➔ [Sievgene in-silicoスクリーニング:作業領域]ダイアログが表示されます。
![[Sievgene in-silicoスクリーニング:作業領域]ダイアログ](../gfx/12.files/image002.png)
各項目を入力します。
|
ホスト名 |
計算サーバーのホスト名またはIPアドレスを入力します。 |
|
ユーザー名 |
計算サーバーにログインするためのユーザー名を入力します。 |
|
パスワード |
計算サーバーにログインするためのパスワードまたはパスフレーズを入力します。 |
|
ポート番号 |
計算サーバーにssh接続するためのTCPポート番号です。ssh用のポート番号は一般的には22ですが、サーバーの手引書や管理者の指示に従ってください。 |
|
screening_dataディレクトリーのパス |
前節「スクリーニングを行う前に」で展開した分子データのフォルダーのパスを指定してください。直下にmts_dataディレクトリーが含まれているはずです。 |
|
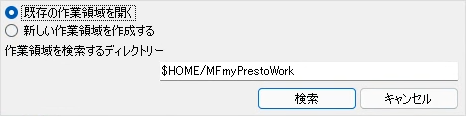
既存の作業領域を開く |
過去に作成した作業ディレクトリーを再利用してスクリーニングを行います。 |
|
新しく作業領域を作成する |
作業ディレクトリーを新たに作成し、その中に初期データをコピーします。 |
|
作業領域を検索するディレクトリー |
過去に作成した作業ディレクトリーの直ぐ上のディレクトリーを指定します。 |
|
作業ディレクトリー |
新しく作成する作業ディレクトリーのパスを指定します。 |
[新しい作業領域を作成する]ラジオボタンにチェックを入れた状態で[作成]ボタンをクリックすると[Sievgene in-silicoスクリーニング:Screening ]ダイアログが表示されます。
[既存の作業領域を開く]ラジオボタンにチェックを入れると[作業領域を検索するディレクトリー]エディットボックスが表示され、[作成]ボタンが[検索]ボタンに変わります。
[検索]ボタンをクリックすると[見つかった作業エリア]ダイアログが表示されます。
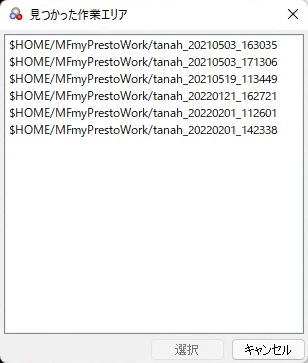
[見つかった作業エリア]ダイアログのリストから再利用する作業ディレクトリーを選んで[選択]ボタンをクリックすると[Sievgene in-silicoスクリーニング:Screening1]ダイアログが表示されます。
▲ 上に戻る
ジョブスケジューラーの選択
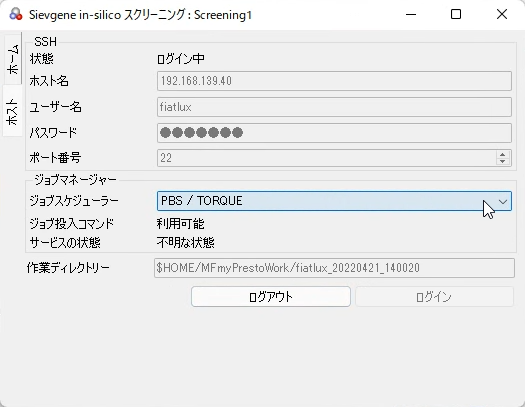
ジョブスケジューラーはデフォルトでは“Slurm”が選択されています。ジョブスケジューラーを変更する場合は以下の手順で行います。
[Sievgene in-silicoスクリーニング:Screening1]ダイアログ左端の[ホスト]タブをクリックしてホスト設定画面を表示させます。[ジョブスケジューラー]コンボボックスで利用したいジョブスケジューラーを選択します。
▲ 上に戻る
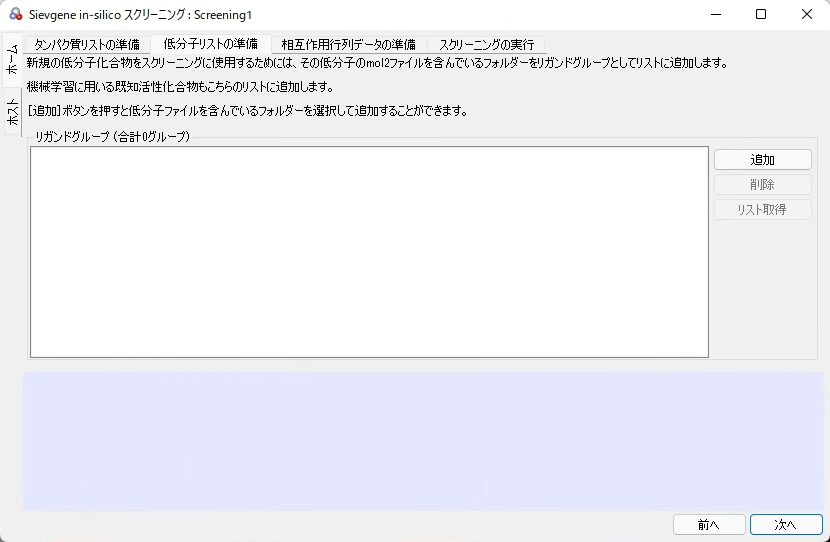
タンパク質リストの準備
[Sievgene in-silicoスクリーニング:Screening1]ダイアログが開くとまず[タンパク質リストの準備]画面が表示されます。[タンパク質リストの準備]画面ではターゲットタンパク質の設定を行います。

ターゲットタンパク質の追加
ターゲットタンパク質を追加する場合は、[タンパク質リストの準備]タブの[追加]ボタンをクリックします。
➔ シーン中から「受容体ポケットの生成」まで終えた状態のタンパク質を探して計算サーバーに転送します。
mm_workspaceファイルに保存したタンパク質をターゲットとして使用する場合は、[追加]ボタンのプルダウンメニューにある“mm_workspaceファイルから追加”をクリックします。するとファイルダイアログが開きますので、ターゲットタンパク質が含まれているmm_workspaceファイルを選択します。
➔ mm_workspaceファイル中から「受容体ポケットの生成」まで終えた状態のタンパク質を抽出して計算サーバーに転送します。
Pro.pdb、Pro.tpl、point.pdbファイルとして保存したタンパク質をターゲットとして使用する場合は、[追加]ボタンのプルダウンメニューにある“pdb・tpl・pocketファイルのフォルダーを追加”をクリックします。するとフォルダー選択ダイアログが開きますので、Pro.pdb、Pro.tpl、point.pdbの3ファイルを含んでいるフォルダーを選択します。
➔ 選択したフォルダーに含まれているPro.pdb、Pro.tpl、point.pdbを計算サーバーに転送します。フォルダー名がタンパク質名になります。
![]() 半角英数字および「-」「_」のみタンパク質名に使用できます。
半角英数字および「-」「_」のみタンパク質名に使用できます。
ターゲットタンパク質が計算サーバーに転送されるとタンパク質リストにタンパク質名が追加され、[次へ]ボタンが有効になります。
![]()
タンパク質リストの項目を選択すると[削除]ボタンと[プレビュー]ボタンが有効になります。
DSI法を行う場合は[DSI法を実行する]チェックボックスにチェックを入れます。
![]()
➔ タンパク質リストが無効になり[次へ]ボタンが有効になります。
▲ 上に戻る
ターゲットタンパク質の削除
タンパク質リストの項目を選択して[削除]ボタンをクリックすると計算サーバー上のタンパク質データを削除します。
![]() この操作は元に戻せません。
この操作は元に戻せません。
▲ 上に戻る
ターゲットタンパク質のプレビュー
タンパク質リストの項目を選択して[プレビュー]ボタンをクリックすると計算サーバー上のタンパク質データを取得してシーンウィンドウに表示します。
ターゲットタンパク質の設定が完了したら[次へ]ボタンをクリックします。
➔ [低分子リストの準備]画面に切り替わります。
▲ 上に戻る
低分子リストの準備
[低分子リストの準備]画面では既知活性化合物や検索対象化合物の設定を行います。
既知活性化合物の追加
既知活性化合物を追加する場合は次の手順で行います。
1.[追加]ボタンをクリックします。
➔ [リガンドグループの追加]ダイアログが開きます。
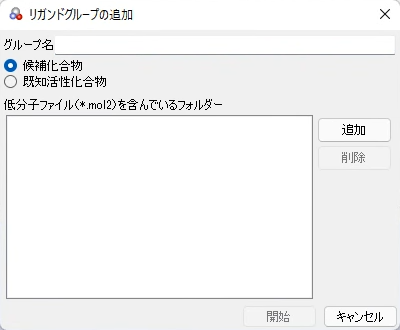
2.[リガンドグループの追加]ダイアログの[既知活性化合物]ラジオボタンにチェックします。
3.[追加]ボタンをクリックするとフォルダー選択ダイアログが開きますので、既知活性化合物のmol2ファイルを含んでいるフォルダーを選択します。このとき同じフォルダーに検索対象化合物が含まれていると、それも既知活性化合物としてまとめますのでフォルダーを分けておいてください。
➔ [グループ名]エディットボックスに選択したフォルダー名が入ります。
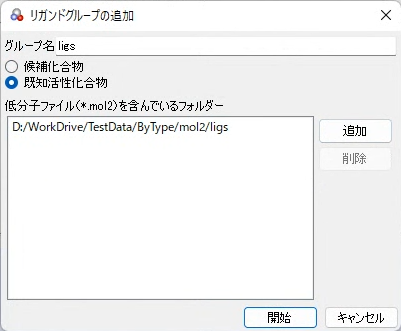
4.必要であれば、[グループ名]エディットボックスでグループ名を変更します。
![]() 半角英数字および「-」「_」のみリガンドグループ名に使用できます。
半角英数字および「-」「_」のみリガンドグループ名に使用できます。
5.[開始]ボタンをクリックします。
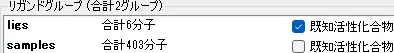
➔ リガンドデータが計算サーバーに転送され[低分子リストの準備]画面のリガンドグループリストにリガンドグループが追加されます。
![]()
▲ 上に戻る
検索対象化合物の追加
検索対象化合物を追加する場合は次の手順で行います。
1.[追加]ボタンをクリックします。
➔ [リガンドグループの追加]ダイアログが開きます。
2.[リガンドグループの追加]ダイアログの[候補化合物]ラジオボタンにチェックします。
3.[追加]ボタンをクリックするとフォルダー選択ダイアログが開きますので、検索対象化合物のmol2ファイルを含んでいるフォルダーを選択します。
➔ [グループ名]エディットボックスに選択したフォルダー名が入ります。
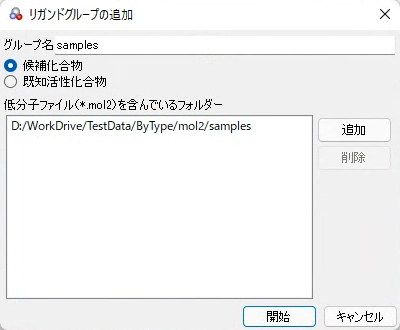
4.必要であれば、[グループ名]エディットボックスでグループ名を変更します。
5.[開始]ボタンをクリックします。
➔ リガンドデータが計算サーバーに転送され[低分子リストの準備]画面のリガンドグループリストにリガンドグループが追加されます。
▲ 上に戻る
低分子化合物の削除
リガンドグループリストの項目を選択して[削除]ボタンをクリックすると計算サーバー上の化合物データを削除します。
![]() この操作は元に戻せません。
この操作は元に戻せません。
▲ 上に戻る
低分子リストの取得
リガンドグループリストの項目を選択して[リスト取得]ボタンをクリックすると選択したリガンドグループに含まれているmol2ファイルのリストを表示します。
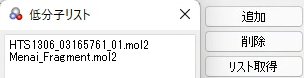
低分子化合物の設定が完了したら[次へ]ボタンをクリックします。
➔ [相互作用行列の準備]画面に切り替わります。
▲ 上に戻る
相互作用行列の準備
[相互座用行列の準備]画面では、グリッドの作成、ドッキング計算、相互作用行列データの作成を行います。
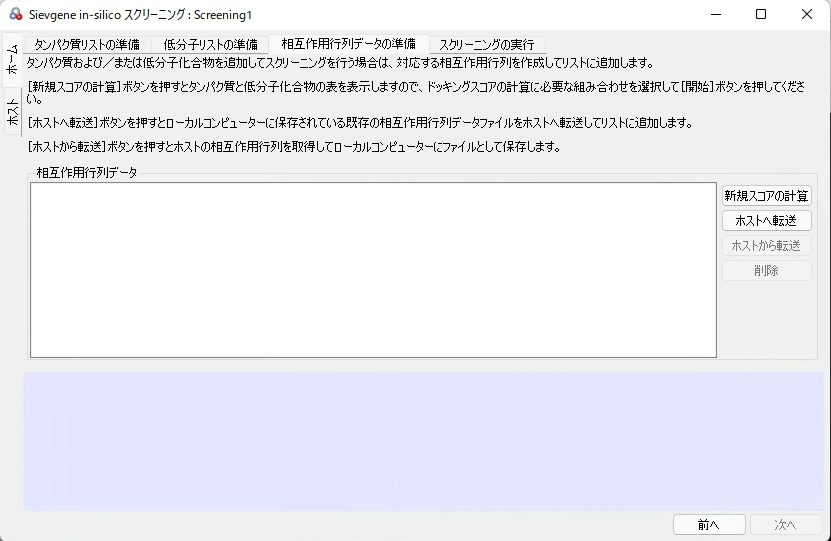
新規スコアの計算
相互作用行列データファイルを新たに作成する場合は次の手順で行います。
1.[相互作用行列の準備]画面の[新規スコアの計算]ボタンをクリックします。
➔ [相互作用行列の計算]ダイアログが表示されます。
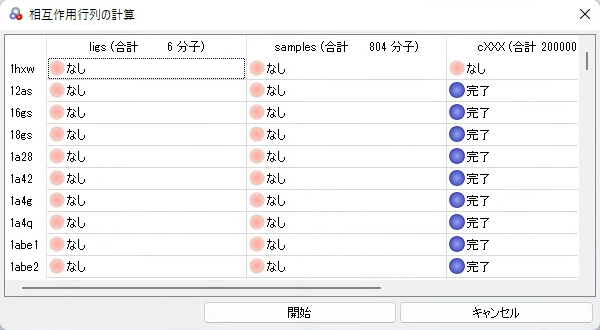
タンパク質(ターゲットタンパク質とリファレンスタンパク質)とリガンドグループの組み合わせで、既に相互作用行列データファイルが作成されていれば“完了”、リガンドグループに含まれる一部の分子とだけ作成されていれば“一部”、全く作成されていなければ“なし”と表示されています。
2.計算を行うタンパク質とリガンドの組み合わせを確認して、[開始]ボタンをクリックします。
➔ 計算が始まります。初回はタンパク質のグリッドを作成してからドッキングスコアの計算を行います。


一度、計算を開始するとEasy myPrestoを終了してもsievgeneの計算は独立して継続されます。
![]() 計算開始後にEasy myPrestoを終了する際は、必ず現在の編集内容をワークスペースファイルとして保存してください。sievgeneのプロセス実行状態を追跡できなくなります。
計算開始後にEasy myPrestoを終了する際は、必ず現在の編集内容をワークスペースファイルとして保存してください。sievgeneのプロセス実行状態を追跡できなくなります。
▲ 上に戻る
相互作用行列データの確認
計算が終了すると自動的にEasy myPresto上に結果がインポートされます。
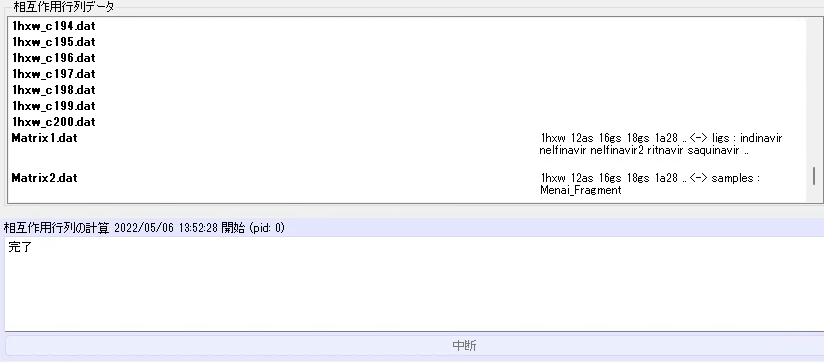
[新規スコアの計算]ボタンをクリックして[相互作用行列の計算]ダイアログを表示させることでタンパク質とリガンドグループの組み合わせについて相互作用行列データファイルの作成が完了していることを確認できます。
相互作用行列データファイルの作成が完了したら[次へ]ボタンをクリックします。
➔ [スクリーニングの実行]画面に切り替わります。
▲ 上に戻る
スクリーニングの実行
[スクリーニングの実行]画面では、スクリーニングのための設定を行い、スクリーニングを開始します。
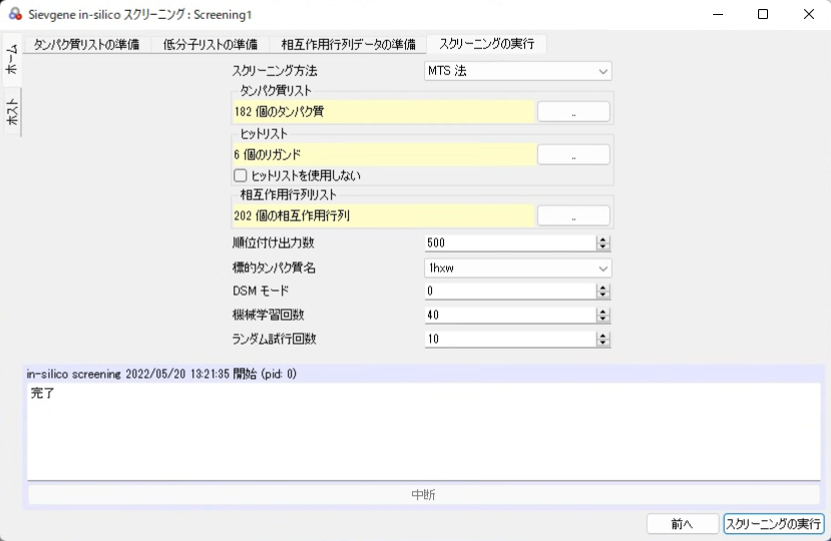
実行
各項目設定・確認し、[スクリーニングの実行]ボタンをクリックします。
|
スクリーニングの方法 |
DSI法かMTS法か選択します。 |
|
タンパク質リスト |
リファレンスタンパク質とターゲットタンパク質が含まれていることを確認します。 |
|
ヒットリスト |
機械学習のための既知活性化合物の分子名が含まれていることを確認します。 |
|
ヒットリストを使用する |
MTS法の場合こちらにチェックを入れるとヒットリストに記載されている分子を機械学習に使用します。チェックを外すと既知活性化合物も候補化合物として扱います。 |
|
相互作用行列リスト |
必要な相互作用行列データファイルが含まれていることを確認します。 |
|
順位付け出力数 |
selectMTSおよびselectDSIプログラムの出力順位数を指定します。 |
|
標的タンパク質名 |
MTS法を行う場合に、ターゲットタンパク質を選択します。 |
|
DSMモード |
MTS法を行う場合に、DSMモードを使用するかどうか指定します。 |
|
機械学習回数 |
selectMTSおよびselectDSIプログラムの機械学習回数を指定します。 |
|
ランダム試行回数 |
selectMTSおよびselectDSIプログラムのランダム試行回数を指定します。 |
▲ 上に戻る
結果の取得
スクリーニングが完了すると自動的に[スクリーニング結果]ウィンドウが表示されます。
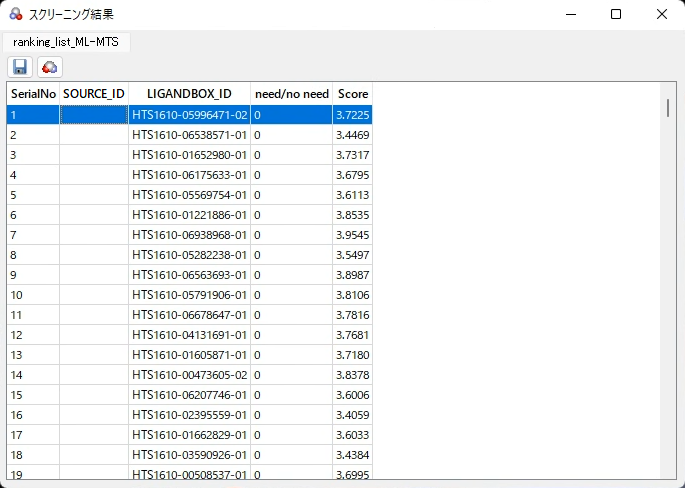
[スクリーニング結果]ウィンドウでは下記の操作が行えます。
|
|
表の項目を選択した状態で糖ボタンをクリックすると、スクリーニング用の分子データから該当する化合物の構造をシーンに読み込みます。 
|
|
|
表のデータをCSV形式でファイルに保存します。 |
ツリービューに"rankink_list_{スクリーニングの方法}"という項目と、DSI法またはMTS法でヒットリストを使用する場合は"removed_from_comp_list"という項目が追加されます。これらの項目を右クリックして[ファイルの書き出し…]を選択すると表のデータをCSV形式でファイルに保存することができます。

▲ 上に戻る