分子データの座標と、トラジェクトリーデータの座標との構造の差をRMSD(Root mean square deviation)として計算します。また、トラジェクトリーデータの構造の揺らぎをRMSF(Root mean square fluctuation)として計算します。手順は以下のとおりです。
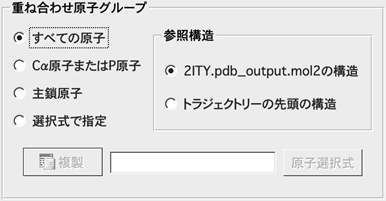
1.構造の重ね合わせに使う原子を指定します。
RMSD・RMSFの計算に先立ってトラジェクトリーの各構造を参照構造へ重ね合わせを行いフィットさせます。重ね合わせに使う原子は[すべての原子]、[Cα原子またはP原子]、[主鎖原子]または[選択式で指定]から選択します。[選択式で指定]を選んで[複製]ボタンをクリックするとシーンの選択状態から選択式を作成します。参照構造は[(分子データ)の構造]または[トラジェクトリーの先頭の構造]から選択します。
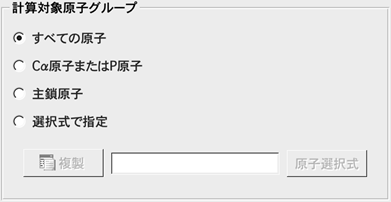
2.RMSD・RMSFの計算に使う原子を指定します。
RMSD・RMSFの計算の対象とする原子を[すべての原子]、[Cα原子またはP原子]、 [主鎖原子]または[選択式で指定]から選択します。[選択式で指定]を選んで[複製] ボタンをクリックするとシーンの選択状態から選択式を作成します。
![]() [原子選択式]ボタンをクリックすると選択式の一覧表が表示されます。
[原子選択式]ボタンをクリックすると選択式の一覧表が表示されます。
![]() 選択式については、「原子選択式」を参照してください。
選択式については、「原子選択式」を参照してください。
3.[RMSDの計算]ボタンまたは[RMSFの計算]をクリックします。

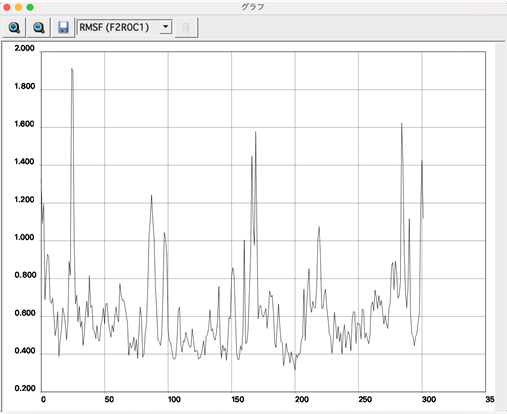
➜ 計算結果を示すグラフが表示されます。
出力結果
以下の内容で結果が表示されます。左上のフロッピーディスクのアイコンのボタンをクリックするとグラフのデータをCSV形式またはグラフの画像をPNG形式でファイルに保存することができます。