9.2. スクリーニング
概要
Note
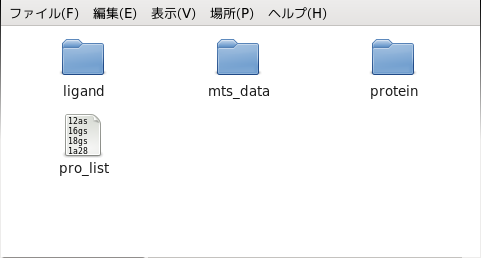
スクリーニング機能には計算用のLinux PCが必要です。また、Slurm等ジョブ管理システムと、スクリーニング用の分子データ(バイオ産業情報化コンソーシアム(JBIC)が配布している下図のデータ)のインストールが必要になります。
図1:スクリーニング用の分子データ
実行手順は下記の通りです。
トポロジーファイルとプローブ点を作成した分子データを用意します。
Note
DSI法は標的タンパク質の構造を使いません。当手順は不要です。
[スクリーニング]ボタンをクリックします。
図2:[スクリーニング]のクリック

計算用PCに接続するためのSSHの設定を入力後、[既存の作業領域を開く]または[新しい作業領域を作成する]を選び、[検索]または[作成]ボタンをクリックします。
図3:[作成]のクリック

- [タンパク質のリストの準備]タブが表示されます。[ホスト]タブのジョブスケジューラーの設定が正しいかご確認ください。[ホーム]タブの[追加]→[OK]をクリックし、タンパク質のデータをホストに転送した後、[次へ]ボタンをクリックします。
図4:[追加]のクリック

Note
DSI法の時は[DSI法を実行する]をチェックし[追加]ボタンは押さずに[次へ]をクリックしてください。
ワークスペースにトポロジーファイルとプローブ点作成済みの同名の分子が複数ある時は[追加]の操作がエラーになります。エラーが出る時は分子名をプロパティビュー等で変更してください。
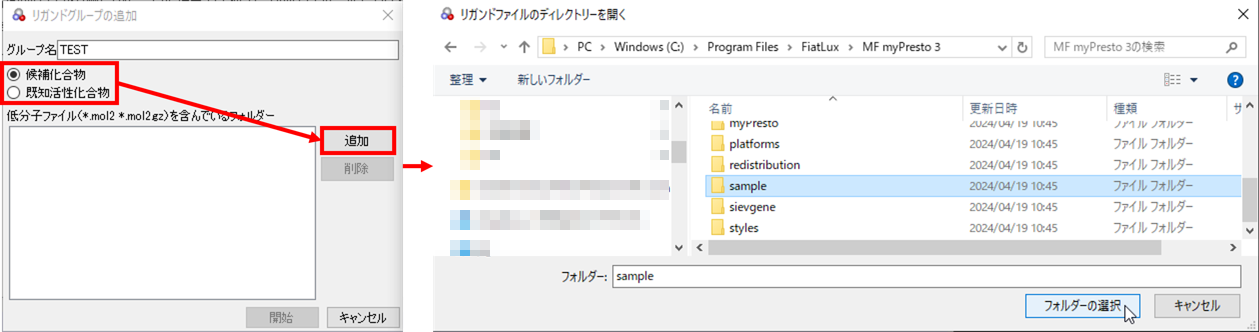
[低分子リストの準備]タブが表示されますので、既存の候補化合物や機械学習用の既知活性化合物がある場合は[追加]ボタンをクリックします。
図5:[リガンドグループの追加]の[追加]ボタンでmol2ファイルがあるフォルダを指定する例

[リガンドグループの追加]ダイアログが表示されますので[候補化合物][既知活性化合物]のいずれかをチェックし、[追加]ボタンからmol2ファイルのあるフォルダを選びます。設定後に[次へ]ボタンをクリックします。
Note
DSI法の時は既知活性化合物を必ず指定します。MTS法では既知活性化合物を指定するとML-MTS法で計算を行います。
[リガンドグループの追加]ダイアログの[グループ名]には任意の名前を設定します。
[相互作用行列データの準備]タブで、[新規スコアの計算]→[開始]の順にボタンをクリックすると相互作用行列の計算を開始します。
図6:[開始]のクリック

Note
計算途中でMF myPrestoを終了する場合、Workspaceファイルとして保存しておくと、次回起動時に計算途中から復帰できます。
計算にはしばらく時間がかかります。計算終了後は自動で[スクリーニングの実行]のタブに遷移します。
[スクリーニング実行]タブでパラメータを設定し、[スクリーニングの実行]ボタンをクリックします。
図7:[スクリーニング実行]のクリック

Note
計算にはしばらく時間がかかります。計算終了後は[スクリーニング結果]のダイアログが表示されます。
[スクリーニング結果]のダイアログのリストで項目を選択し、
 のボタンをクリックすると、構造と化合物のIDをワークスペースに読み込みます。[スクリーニング結果]のダイアログが非表示の場合は[スクリーニング]タブの[結果]ボタンから表示できます。
のボタンをクリックすると、構造と化合物のIDをワークスペースに読み込みます。[スクリーニング結果]のダイアログが非表示の場合は[スクリーニング]タブの[結果]ボタンから表示できます。図8:スクリーニング結果のダイアログの例

Note
化合物のIDを使ってナミキ商事社やキシダ化学社から実際の試薬を購入できます。