分子の重ね合わせ配置は、指定した2つの分子のRMSD (root-mean-squared deviation) が最小となるように重ね合わせ処理をおこなうものです。
[タンパク質編集]タブの[分子の重ね合わせ配置…]ボタンをクリックします。

➜ [分子の重ね合わせ配置]ダイアログが表示されます。
各部の名称
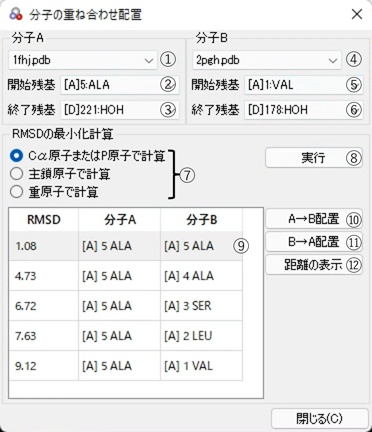
|
|
名称 |
説明 |
|
① |
分子A選択ボックス |
重ね合わせの対象となる分子です。重ね合わせができる分子は、タンパク質分子または核酸分子です。分子Aと分子Bは同一の分子データは指定できません。 |
|
② |
分子A開始残基選択ボックス |
重ね合わせ処理を開始する、分子Aの残基を選択します。分子A選択時は、シークエンス内の最初の残基が設定されています。 |
|
③ |
分子A終了残基選択ボックス |
重ね合わせ処理を終了する、分子Aの残基を選択します。分子A選択時は、シークエンス内の最後の残基が設定されています。 |
|
④ |
分子B選択ボックス |
重ね合わせの対象となる分子です。 |
|
⑤ |
分子B開始残基選択ボックス |
重ね合わせ処理を開始する、分子Bの残基を選択します。分子B選択時は、シークエンス内の最初の残基が設定されています。 |
|
⑥ |
分子B終了残基選択ボックス |
重ね合わせ処理を終了する、分子Bの残基を選択します。分子B選択時は、シークエンス内の最後の残基が設定されています。 |
|
⑦ |
比較原子指定ラジオグループ |
下記RMSD計算実行時に使用する原子を選択します。[Cα原子またはP原子で計算]は、タンパク質分子の場合Cα原子、核酸分子の場合はリン原子のみを使用します。[主鎖原子で計算]は、タンパク質分子、核酸分子の主鎖原子を使用します。[重原子で計算]は、重原子のみを使用します。[重原子で計算]は、分子のシークエンスが異なると計算できません。 |
|
⑧ |
RMSD計算実行 ボタン |
分子Aと分子Bの開始残基から終了残基までのシークエンスを用い、配置の結果、最小となるRMSD値を計算し、[RMSD値]リストに結果を表示します。分子Aと分子Bとで残基の数が異なる場合は、残基の数が少ない方を、多い方に対してシークエンスをシフトしながら順番にRMSD値を計算します。 |
|
⑨ |
RMSD値リスト |
RMSD値の計算結果が表示されます。結果は、RMSD値が小さい順にソートして表示されます。[RMSD値]リスト中のカラム[分子A]および[分子B]は、RMSD計算をおこなった対の各先頭残基名です。計算直後はRMSD値が最小の対が選択されています。 |
|
⑩ |
分子A配置ボタン |
分子Aを分子Bに対し配置します。配置は、[RMSD値]リストの選択されている結果をもとに行われます。 |
|
⑪ |
分子B配置ボタン |
分子Bを分子Aに対し配置します。 |
|
⑫ |
距離の表示ボタン |
分子Aと分子Bの配置の結果、RMSD値が最小となるときの対応する各原子間の距離を表示します。 |
|
⑬ |
閉じるボタン |
ダイアログを閉じます。 |
操作の手順
ここでは、実際に分子ファイルを読み込んで、重ね合わせをおこなう手順を紹介します。
1.PDBファイル”1fhj.pdb”および”2pgh.pdb”を読み込みます。
2.[タンパク質編集]タブの[分子の重ね合わせ配置…]ボタンをクリックします
➜ [分子の重ね合わせ配置]ダイアログが表示されます。
3.[分子A選択]ボックスから、分子名”1fhj.pdb”を選択します。
4.[分子B選択]ボックスから、分子名”2pgh.pdb”を選択します。
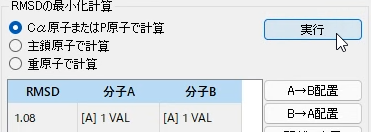
5.[RMSD計算実行]ボタンをクリックします。
➜ [RMSD値]リストに重ね合わせ配置のための計算結果が表示されます。
この例では、”1fhj.pdb”および”2pgh.pdb”の残基数が同じであり、開始残基、終了残基も同じため、結果は1つのみ求まります。
6.[距離の表示]ボタンをクリックします。
➜ 分子Aと分子Bの対応する各原子間の距離が表示されます。
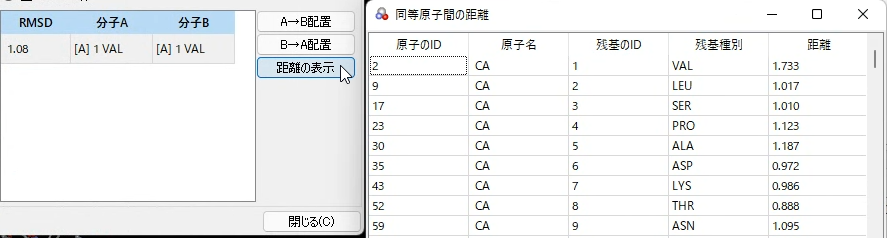
7.[分子A配置]ボタンをクリックします。
➜ 分子Aである、”1fhj.pdb”が分子Bの”2pgh.pdb”に対して配置されます。
