ドッキング計算のための設定をおこないます。
設定は、[設定]タブからおこないます。
設定可能な項目は以下のとおりです。
計算方法
![]()
計算方法を以下から選択します。
|
Fast (実行時間最優先) |
実行時間を最優先する計算方法です。そのため計算精度は低くなります。この計算方法は、以下のカスタム設定に対応します。
|
||||||||||||||||||||||||||||||||||||||
|
Moderate (実行時間、精度重視) |
実行時間、ドッキング精度とも重視する計算方法です。この計算方法は、以下のカスタム設定に対応します。
|
||||||||||||||||||||||||||||||||||||||
|
Precise (精度最優先) |
ドッキング精度を最優先する計算方法です。この計算方法は、以下のカスタム設定に対応します。
|
||||||||||||||||||||||||||||||||||||||
|
カスタム設定 |
カスタム設定をおこないます。 詳細は、後述のとおりです。
|
||||||||||||||||||||||||||||||||||||||
コンフォーマー:原子モデル
![]()
原子モデルを以下から選択します。ドッキング精度を保つためには、“all-atom”を選択してください。
|
united-atom |
|
all-atom |
コンフォーマー:生成する配座数
![]()
生成する配座数を設定します。
sievgeneではこの個数分の配座のドッキングを試した後、もっともらしいドッキング位置に探索空間を絞り、再度この数分だけ異なる配座を発生してドッキングをおこなう操作を後述の“絞込み回数”分だけおこないます。
コンフォーマー:原子間距離のDamping factor
![]()
van der Waals半径のダンピングファクターを設定します。
分子内衝突判定後に原子間衝突のある配座を除外する際に利用されます。
コンフォーマー:トーション回転候補の個数
![]()
配座を生成する際のtorsion回転の候補数を設定します。
2面角は“360/候補の個数“の回転角で回転します。
ドッキング(1):ドッキング方法
![]()
ドッキングの方法を以下から選択します。
|
フレキシブル |
リガンドをフレキシブルにします。
|
|
剛体 |
リガンドを入力配座に固定します。
|
ドッキング(1):ドッキングスピード
![]()
タンパク質分子とリガンドの重ね合わせの際に、重ね合わせの対象外となる原子を計算対象から除外するかしないかを以下から選択します。
|
高速 |
除外します。
|
|
通常 |
除外しません。
|
ドッキング(1):ローカルサーチ対象数
![]()
ローカルサーチの対象数を設定します。
ドッキング(1):絞込みの回数
![]()
“コンフォーマー:生成する配座数”を参照してください。
ドッキング(1):リガンドの複合面三角形数
![]()
ドッキングの際にリガンドの接合面を選択する個数を設定します。
sievgene では接合面を3原子が構成する三角形と規定しています。
ドッキング(1):三角形の原子タイプの適合度
![]()
sievgeneでは原子の特性により接合に関連するかどうかの重みを設定しています。
この適合度を調整することで、不要な接合判定が減少しドッキング計算時間を減少させることができます。1~5までの値が設定でき、値が大きいほど接合面が限定されます。
ドッキング(1):三角形の辺の長さの下限側
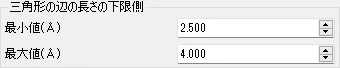
Hash表と比較する原子間距離の下限の最小値と最大値を設定します。
sievgene では接合面を3原子が構成する三角形と規定しています。ここでの設定は、接合面の数を満たす三角形の辺の長さを指定することになります。与えられた辺の長さの範囲から接合面の個数を満たす辺の長さを計算し、接合面を決定します。
ドッキング(1):三角形の辺の長さの上限側
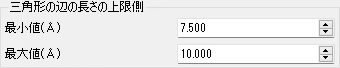
Hash表と比較する原子間距離の上限の最小値と最大値を設定します。
ドッキング(2):スコア計算の重み
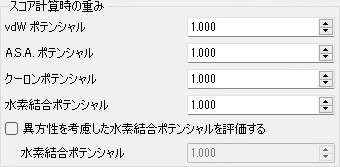
sievgeneでのスコアは、A.S.A.、coulomb、水素結合、異方性を考慮した水素結合、van der Waalsの5つのポテンシャルの和で表現されます。各ポテンシャルに重みを設定したい場合に利用します。
ドッキング(2):リガンドのOH基を回転する
![]()
ドッキング計算時の、リガンド-OH基の末端を回転するかどうかを設定します。
ドッキング(2):タンパク分子の側鎖を回転する
![]()
ドッキング計算時の、タンパク質分子の水素結合可能な側鎖の回転を設定します。
ドッキング(2):リガンドの座標をずらす回数
![]()
ドッキング計算後、リガンド座標を微調整する際に、座標をずらす回数を設定します。
ドッキング(2):計算時間の上限
![]()
リガンド1分子あたりのドッキング計算時間に上限を設定します。制限時間を越えると計算を打ち切り、途中の計算結果を記録します。
出力:上位スコア出力座標数
![]()
取得する上位スコア座標の個数を設定します。
出力:上位スコア出力数
![]()
取得する上位スコアの個数を設定します。