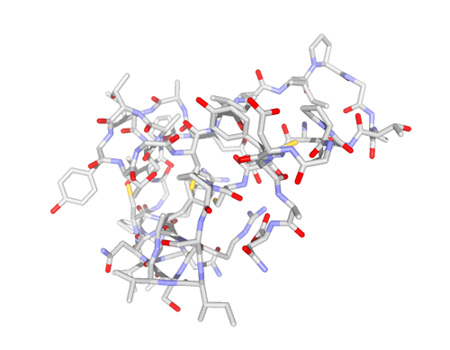
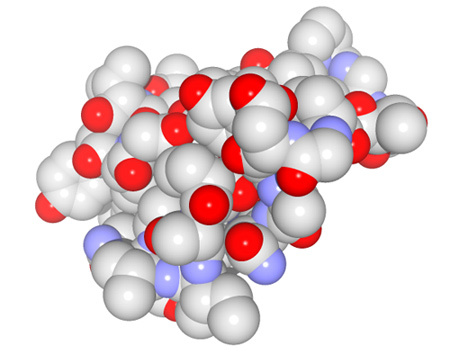
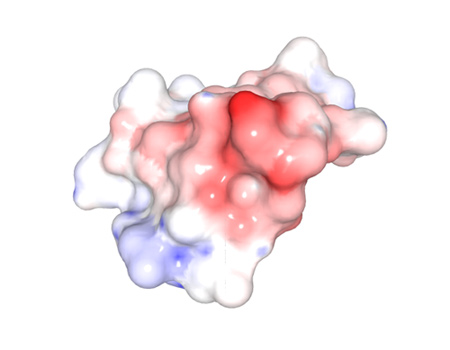
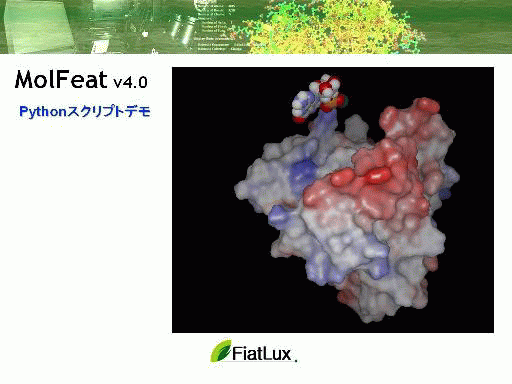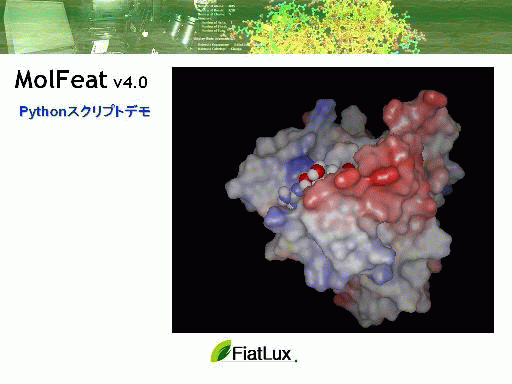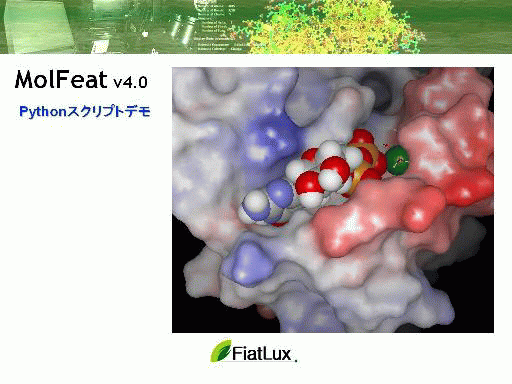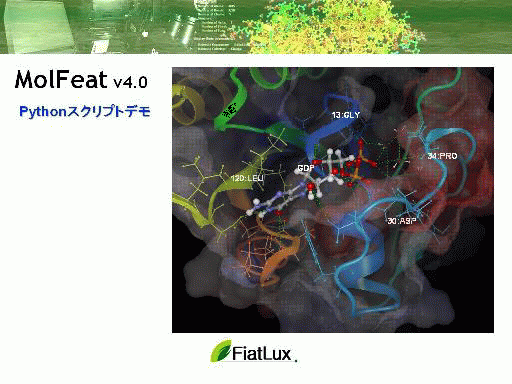
製品概要
MolFeatはタンパク質/核酸のための3次元イメージ作成ソフトウェアです。分子データの読み込みや表示形状の編集、高品質な画像/動画の作成が簡単に行えます。付属のプラグインソフトを使うと、編集した構造をPowerPointのスライド上で、マウス操作により自由に回転/拡大/縮小できます(Windows版のみ対応)。
また、Pythonスクリプトをサポートしており、操作内容をスクリプトファイルに残して繰り返し実行したり、プレゼンテーション用にアニメーションするスクリプトを作成できます。
評価版
下記からMolFeatの評価版をダウンロードいただけます。
ダウンロードはこちらカタログ
下記からMolFeatのカタログをダウンロードいただけます。
ダウンロードはこちらファイルのサポート
分子データの読み込み/保存
- Macromolecular Transmission Format形式 .mmtf
- Macromolecular Crystallographic Information File形式 .cif
- Protein Data Bank形式 .pdb
- MDL社 分子ファイル形式 .mol
- Tripos社 分子ファイル形式 .mol2
トラジェクトリデータの読み込み
- AMBER Parameter/Topology ファイル形式 .parmtop
- AMBER Trajectory ファイル形式 .nc , .mdcrd
- AMBER Restart ファイル形式 .restart
- CHARMM、NAMD形式 .dcd
- TINKER形式 .arc
- GROMACS形式 .trr
- cosgene形式 .cor
分子軌道データの読み込み
- Gaussian 形式 ,cube
電子密度データの読み込み
- .CCP4
- .BRIX
- .CNS
- .SITUS
シーケンスの書き出し
- FASTA形式
- 主鎖2面角φ、ψのファイル出力 ( gnuplot などでプロット可能な形式 )
分子編集機能
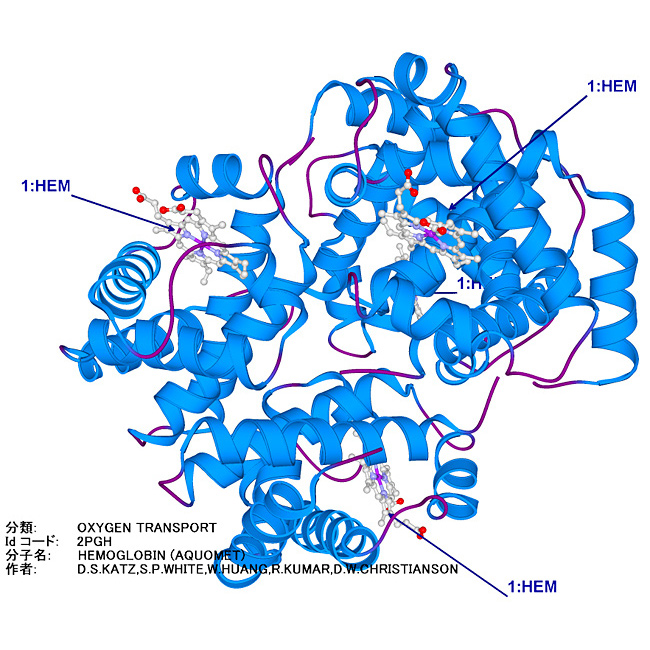
MolFeatはPDBなどの分子構造のデータを読み込み、表現形状や色などを原子単位で自由に編集できます。
また、分子の重ね合わせやポイントミューテーションなどの編集も可能です。
スクリプト実行機能

ご注意ください
上図はGIFで作成したものです。
Pythonスクリプトを使ってMolFeatを操作できるようになりました。ユーザーインターフェースからできる操作とほぼ同等の操作がスクリプトより可能です。 またユーザーインターフェースに対して行った操作を記録し、その内容をPythonスクリプトとして出力することもできます。
例えば以下のようなことができます。
-
操作の自動化
データダウンロード→原子選択→形状編集→高品質イメージ出力などの一連の操作をスクリプトで記述し操作を自動化。
- 分子のアニメーション。
アニメーション機能
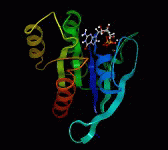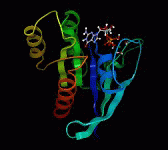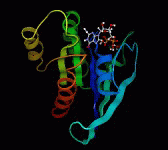
トラジェクトリアニメーションが可能です。PowerPointスライド上でも同様にアニメーションができます。
- トラジェクトリアニメーション
- 物体を中心とした視点の自動回転
- アニメーションスピードの設定
ご注意ください
上図はGIFで作成したものです。
分子軌道表示機能
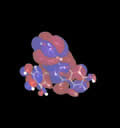
表示された分子構造の上に、分子軌道を重ねて表示できます。
- 表示する分子軌道の選択
- コンター・サーフィス・ドット
- 閾値、色、線幅の設定
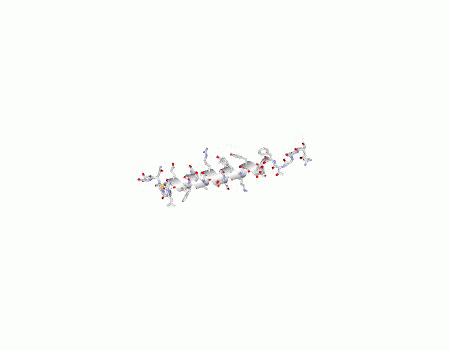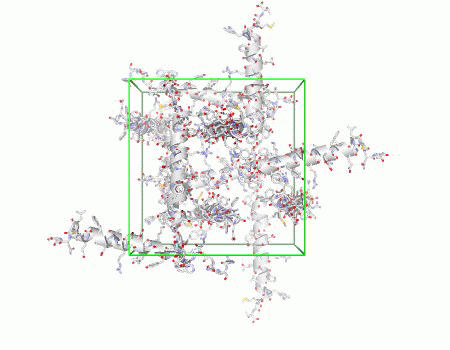
電子密度表示機能
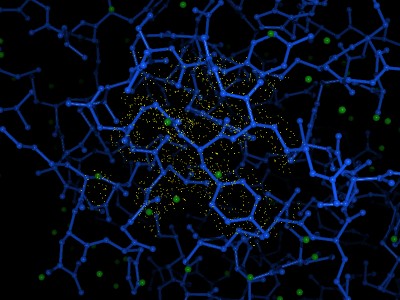
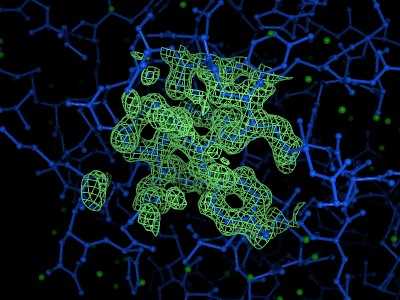
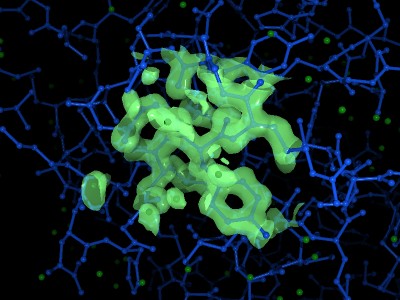
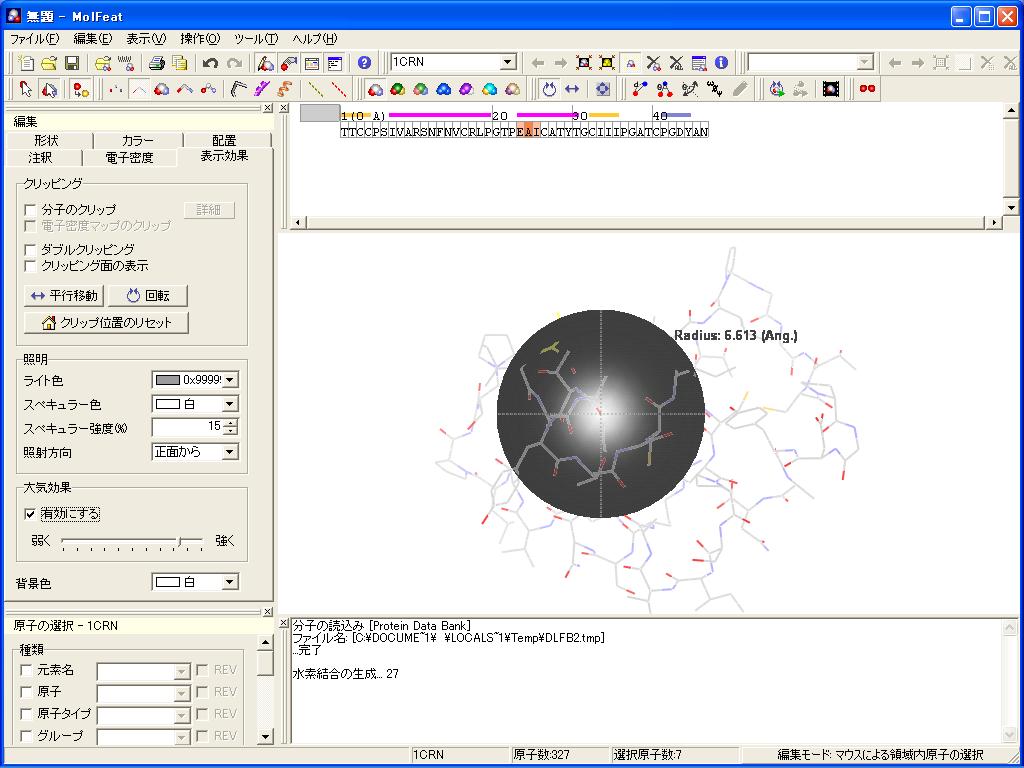
表示された分子構造に、電子密度の等値面を重ねて表示できます。選択された複数の原子周り(残基周り)の電子密度の表示が可能です。またクリックした原子を中心に任意の半径で指定した領域を表示することも可能です。
- 指定原子を中心に表示、複数の原子周りでの表示
- 表示領域、色、線幅の設定
- コンター、ドット、サーフェース表示
- 結晶の表示
注釈機能
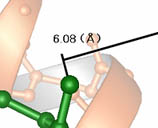
注釈を3次元空間上にマウスを使って自由に配置できます。注釈機能は日本語表示に対応しています。
- 矢印、円弧付きの注釈生成
- 距離などの計測結果の注釈生成
- 上付き下付き文字の表現
プレゼンテーション
機能


編集したデータをもとに、プレゼンテーションのための専用データを作成できます。作成したプレゼンテーションファイルは、付属のプラグインソフトを使用することで、インタラクティブな3次元ビュワーとしてPowerPointのスライドショーやWEBブラウザ上に貼り付けることができます。スライドショー実行中は、マウスを使って自由に分子構造を回転、拡大・縮小できます。(当機能はWindows版のみ対応しております)
高品質イメージ出力
機能
編集した内容をイメージデータとして出力できます。高品質イメージ出力では、ジャギーが少なく高解像度イメージが得られます。学会誌や論文などの印刷物掲載用に最適です。
- ファイルへ出力(BMP、JPG、PNG、TIFF)
- ステレオグラム出力(平行法、交差法)
- レイトレーシング法による高品質イメージ出力
高品質イメージ出力 sample
ステレオグラム(平行法)
ムービーファイル作成機能
操作中の画面をキャプチャした、ムービーファイル(AVIファイル)を作成できます。
ステレオ表示機能

各種ステレオ表示に対応しています。
- フレームシーケンシャル立体表示
- 上下2分割立体表示
- SHARP裸眼立体LCD(LL-151D)
- サイド バイ サイド
- 液晶立体ディスプレイ(Xpol方式)
- zSpace(オプション)
動作環境
Windows版
| OS | Windows 10/11 |
|---|---|
| CPU | Intel Core i 以上 |
| メモリ | 4GB以上 |
| ビデオカード | OpenGL対応ビデオカード QuadBufferを備えたビデオカード(※2) ステレオ表示機能を持ったビデオカード(※2) |
Mac版
| OS | MacOSX 10.13 以降 |
|---|---|
| CPU | Intel Core i 以上 |
| メモリ | 4GB以上 |
| ビデオカード | OpenGL対応ビデオカード |
ご注意ください
※1.PowerPointにMolFeatの画面を貼り付けるプラグインソフト(MolFeatコントロール)はWindows版のみ対応しています。
※2.フレームシーケンシャル方式(NVidia 3D Vision/zSpaceなど)の立体視を行う場合は、上記のQuadBuffer/ステレオ表示機能を備えたビデオカードが必要です。
※3.カタログ記載内容は予告なく変更することがあります。あらかじめご了承ください。
ギャラリー
MolFeatによって作成したプレゼンテーションのサンプルデータをWindowsのPowerPointのスライドショーで3次元表示できます。
MolFeatで編集した分子構造をスライド上でマウスを使って自由な角度から見ることができます。
サンプルデータを正しく表示するには、プラグイン(MolFeatコントロール)をダウンロードし、インストールする必要があります。
MolFeatプラグインのダウンロードはこちらサンプルの実行手順
zipに含まれているpptファイルを開きます。

[セキュリティの警告]が表示されますので[コンテンツの有効化]をクリックします。

PowerPointの[スライドショー]のメニュー(またはF5キー)でスライドショーを実行します。

Pythonスクリプト
ライブラリ
MolFeat上で使用できる、便利なPythonスクリプトのライブラリです。ご自由にダウンロードしてご利用ください。
ベクトル場
表示用サンプル
ベクトル場のファイルを読み込んで、それを矢印注釈で表現します。
ベクトル場のファイルフォーマットは、
vx vy vz dx dy dz
:
vx, vy, vz - ベクトルの開始位置(原子の座標)
dx, dy, dz - ベクトルの向き です。
ファイルは、1GCN.pdb のものです。スクリプトの呼び出し方法は以下のとおりです。
python sample_ana_vec.py
ダウンロードはこちらCA原子しかないPDBで
バックボーンを表示するサンプル
現在読み込まれている分子データをもとに、原子とその次の原子の結合を作るCONECTレコードを生成します。
PDB ファイルのフォーマットに従います。
スクリプトの呼び出し方法は以下のとおりです。
python connect_CA.py <output filename>
仕様手順は以下のとおりです。
・CA 原子しかないPDB ファイルを読み込む
・このスクリプトを実行
・出力されたファイルをオリジナルPDB のATOM またはHETATM 以降に追加
このデータを読み込んでスティック表示にすると、バックボーンスティックのような表示になります。
ダウンロードはこちら新機能・修正点
MolFeat v5.4.0.33
新機能・修正点
MolFeatV5.4.0.33の新機能・修正点は以下のとおりです。
- シークエンスビューの文字サイズ/フォントを変更する機能を追加。
(シークエンスビューを右クリックし[シークエンスビュー]>[フォント設定]から変更できます) - 静電ポテンシャルの各原子の電荷に使う力場を最新の力場に更新。
(力場の詳細はMolFeatの[ヘルプ]>[静電ポテンシャル計算について]を参照) - Appleシリコンに対応。
- MolFeatコントロールの不具合の修正。
MolFeat v5.3.1.32
修正点
MolFeatV5.3.1.32の修正点は以下のとおりです。
- MolFeatコントロールのあるスライドから別のスライドに遷移またはスライドショーを終了してから再表示するとPowerPointが強制終了する不具合を修正。
- MolFeatSimpleViewerでpythonスクリプト実行すると強制終了するバグを修正。
- 旧バージョンのfmpファイルを読み込めない不具合を修正。
MolFeat v5.3.0.31
新機能・修正点
MolFeatV5.3.0.31の新機能・修正点は以下のとおりです。
- MMTFのインポーター、エクスポーターを追加。
- Python2から3に移行。
- MolFeatコントロールの不具合の修正。
- 4Kの画面に表示されるツールボタンが小さい不具合の修正。
- PDBの読み込みで、残基名が変わっても残基番号が変わらなければ同じ残基として認識される不具合の修正。
- その他細かい不具合を修正。
MolFeat v5.2.4.29 修正点
- iMac (Retina 4K, 21.5インチ, 2019)にてシークェンスビューの文字の領域が黒く塗りつぶされる不具合を修正。
- 分子のサーフィスの設定にて断面のオプションが動かない不具合を修正。
- 視点位置の保存機能で、視点を保存後、MolFeatを再起動したときに表示されるデータがおかしい不具合を修正。
- 原子グループの重ね合わせ機能で、ボタンをクリックしているうちに稀にフリーズする不具合を修正。
- ムービーファイルの保存にて、最初のフレームにシークェンスビューの画像が表示される不具合を修正。
- DNA/RNAのmmCIFのファイルを読み込むと結合がおかしくなる不具合を修正。
- 分子軌道の編集でUndo/Redoを繰り返したときに強制終了する不具合を修正。
- Windows版でリボン表示の時にTrajectryの再生が遅くなる不具合を修正。
- PowerPointのスライドショーで、MolFeatコントロールの画面の位置がずれる場合がある不具合を修正。
- その他細かい不具合を修正。
MolFeat v5.2.3.28 修正点
- ユーザーフォルダが非ASCII文字(日本語など)の時に強制終了する不具合を修正。
- httpsのサイトのMolFeatコントロールを表示できない不具合を修正。
- MolFeatコントロールでPowerPointのスライドショーを2回以上開始した場合、2回目以降自動回転が機能しない不具合を修正。
MolFeat v5.2.2.27 修正点
- ポイントスプライトがONの時に、ポイントスプライトに非対応のグラフィックスボード搭載PCで、MolFeatが強制終了する不具合を修正。
MolFeat v5.2.1.26
新機能・修正点
MolFeatV5.2.1.26の新機能・修正点は以下のとおりです。
1. PDBのREMARK 290、3100を使って対称分子を生成する機能を追加。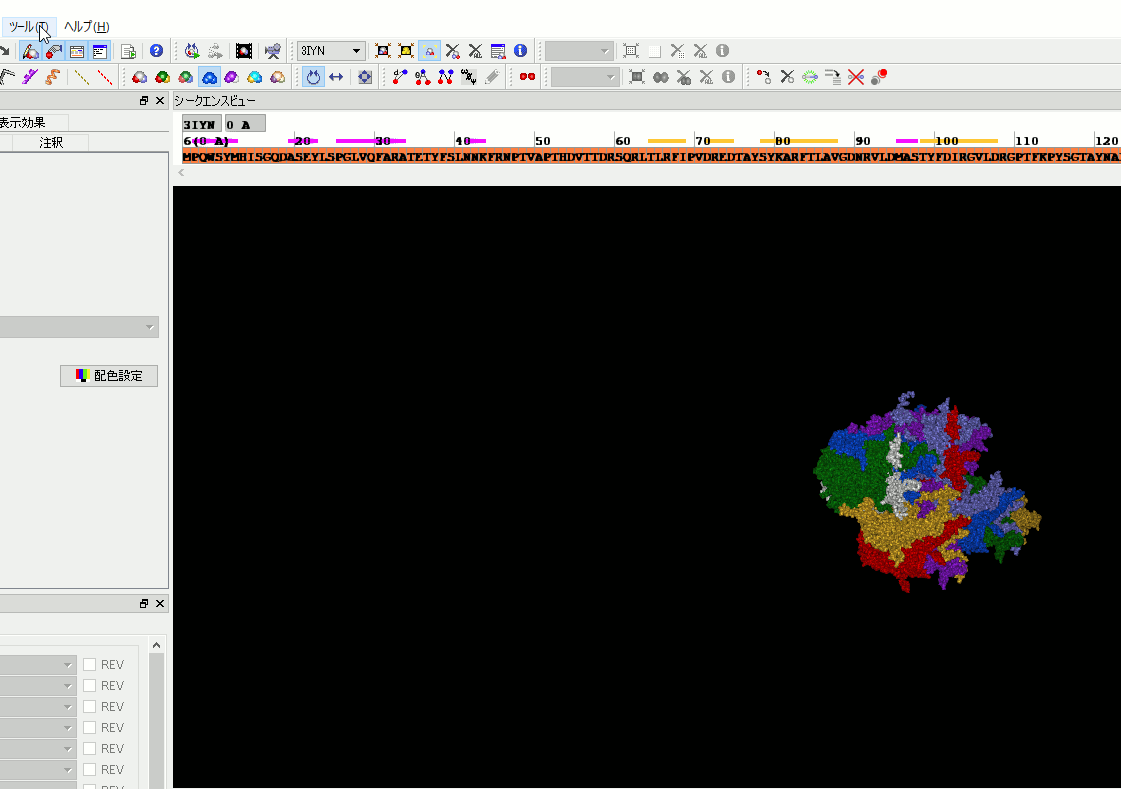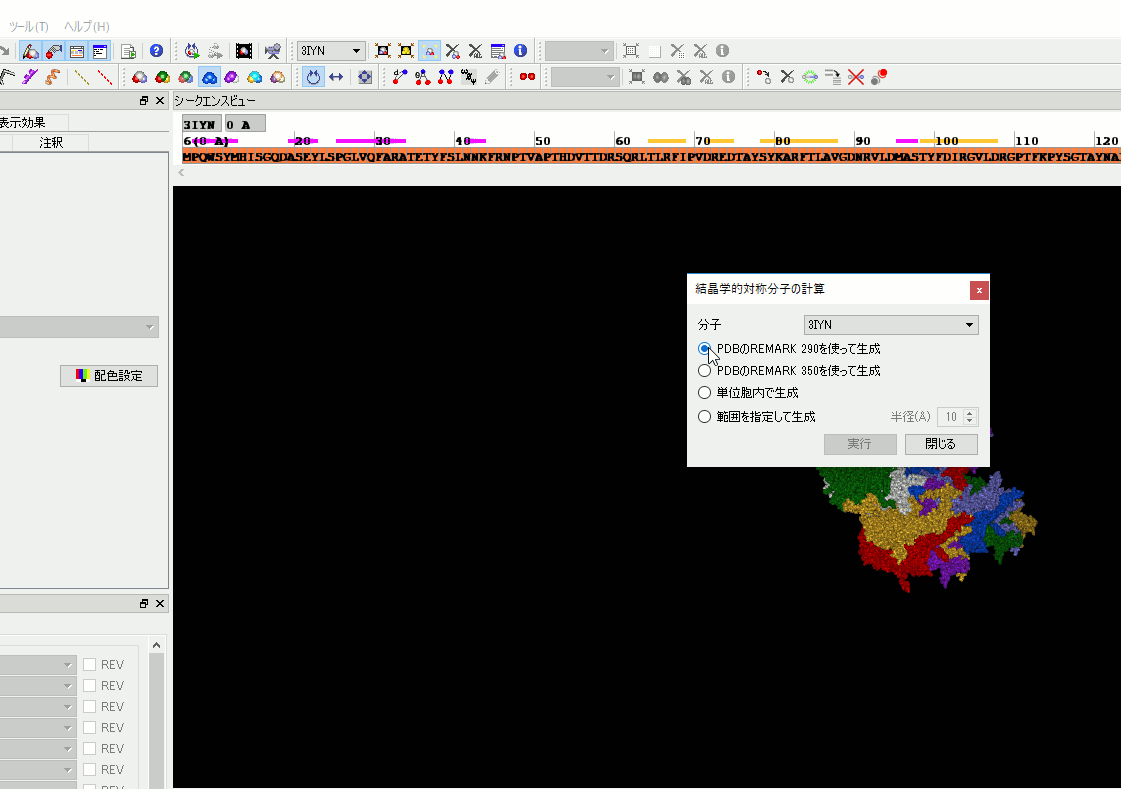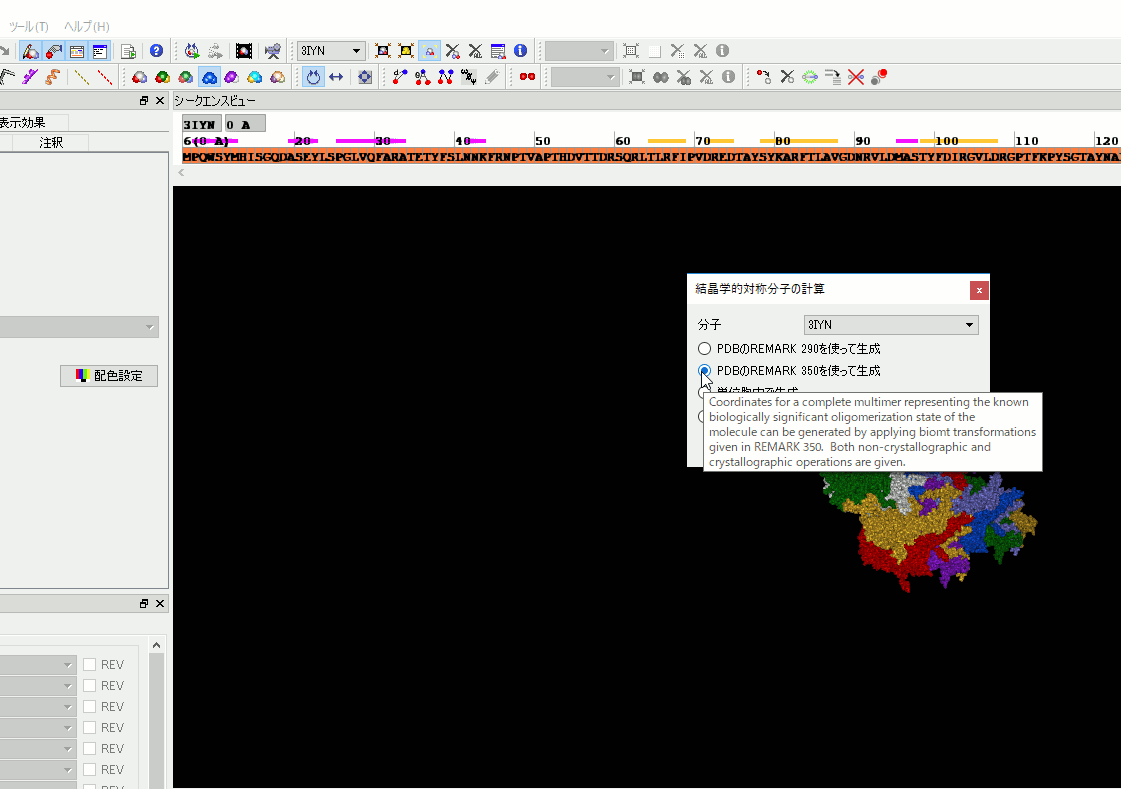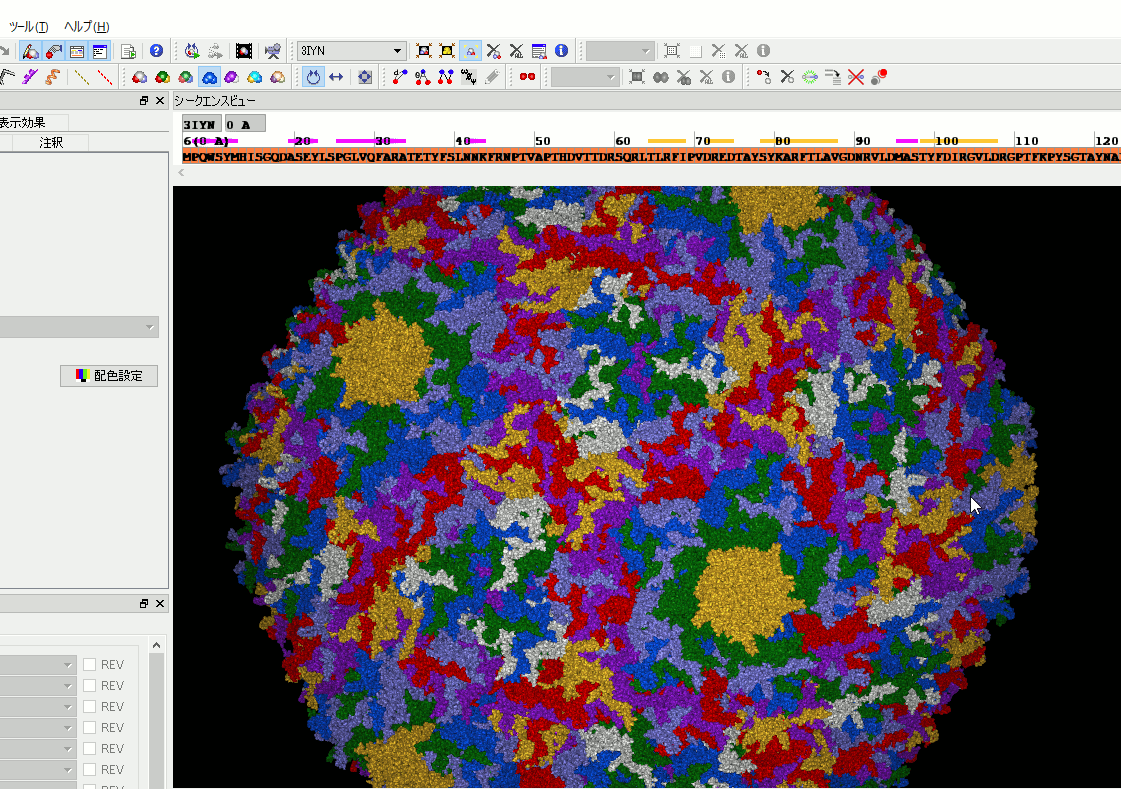
2. 結合を追加・削除する機能を追加。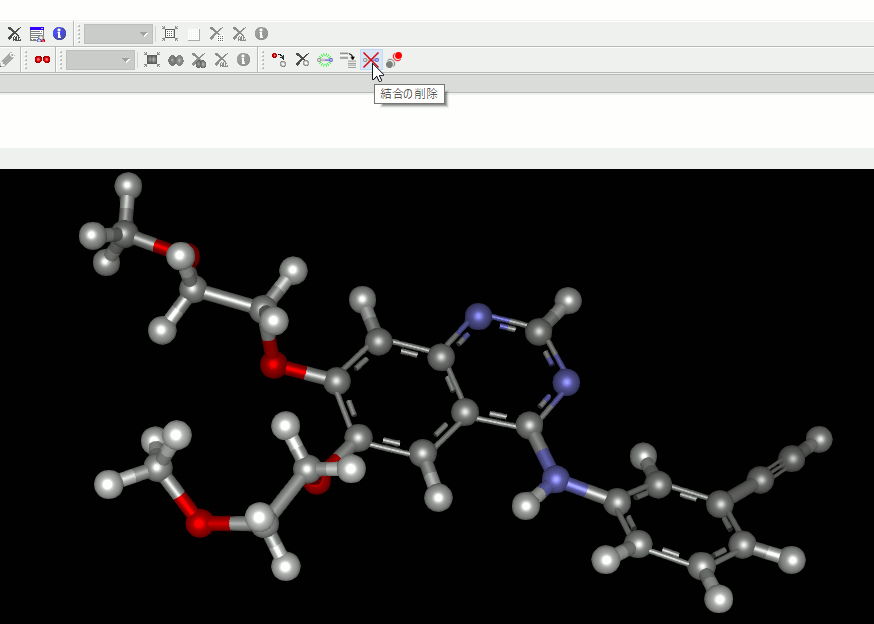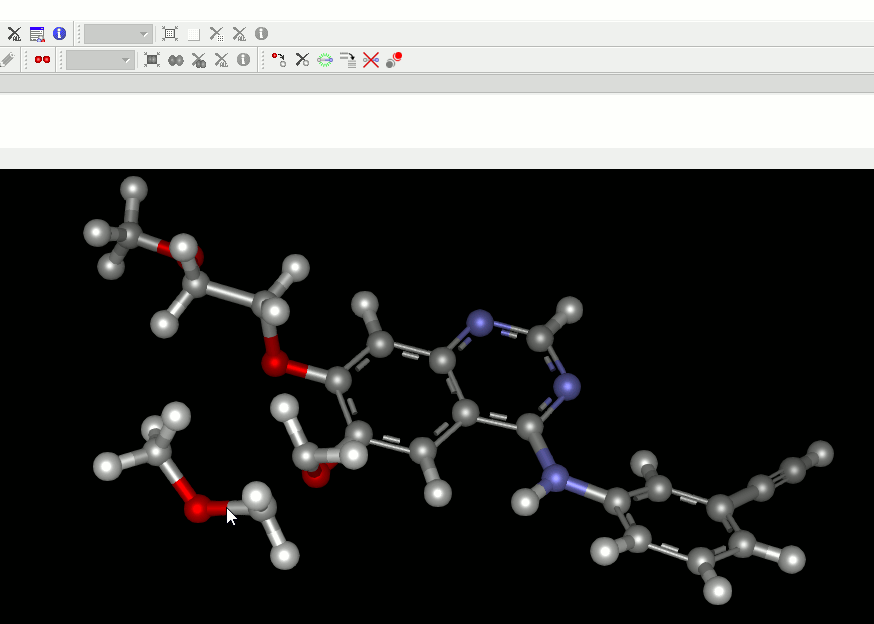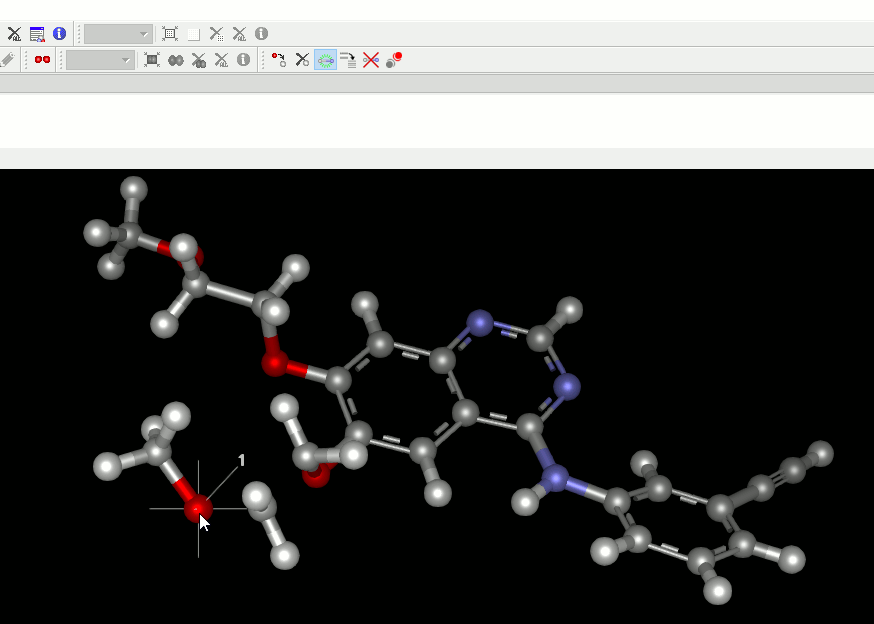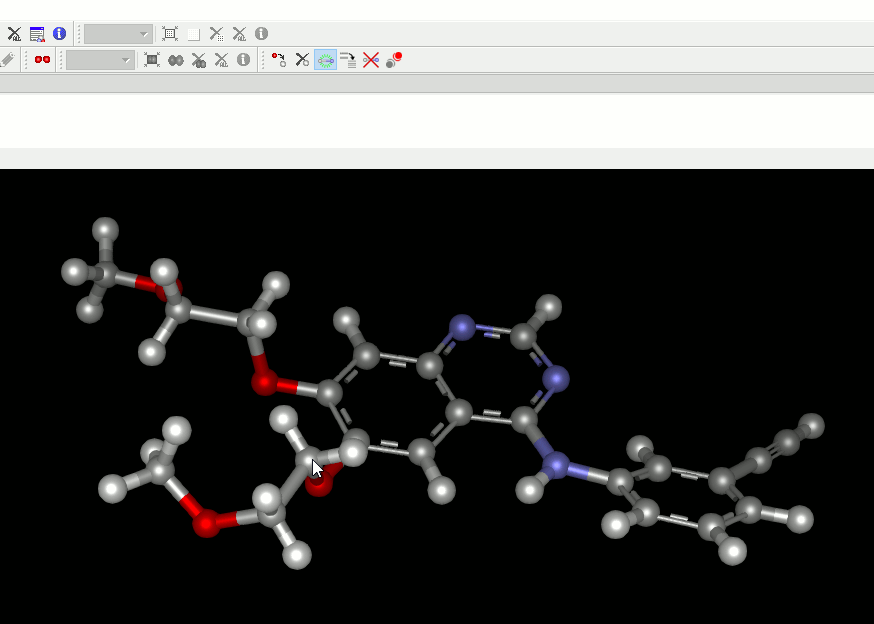
3. 電子密度マップデータの数値精度を向上。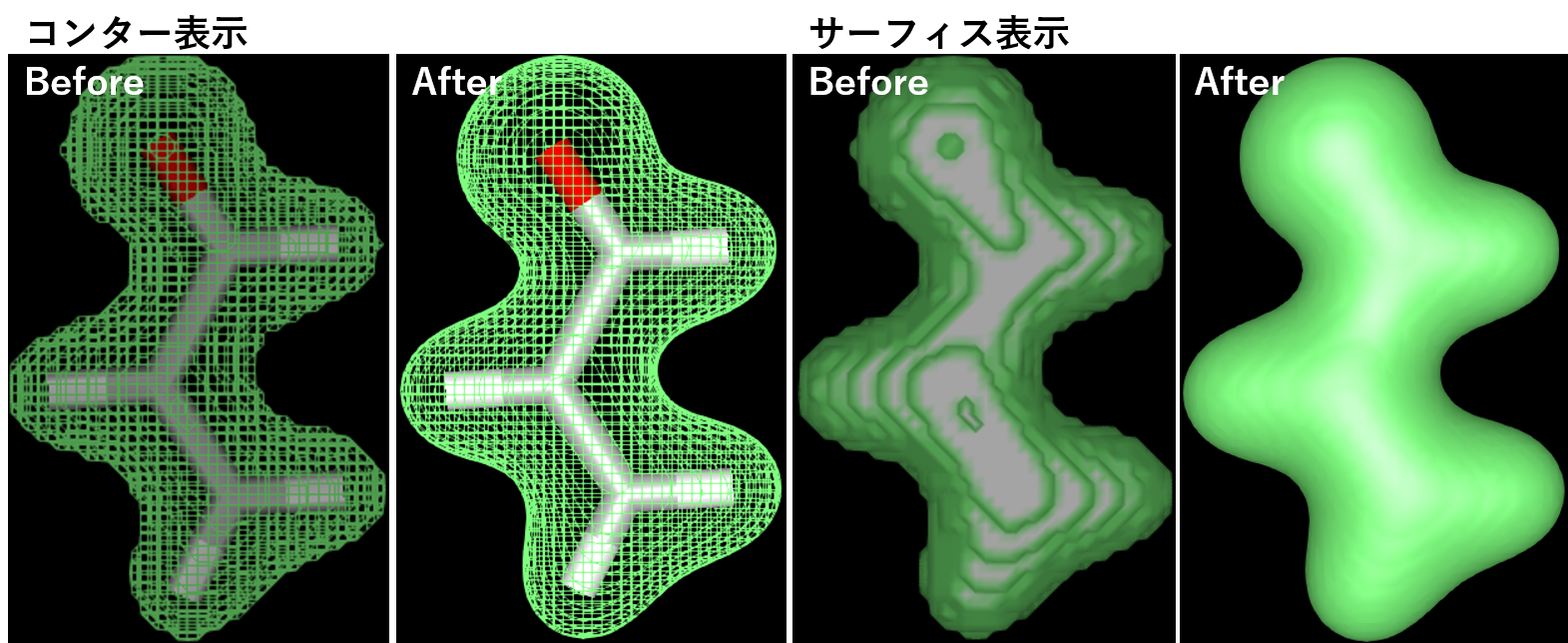
4. Stick表示をpoint spriteで描画し、表示速度を高速化。![]()
5. pngで高品質イメージ出力を保存する際、背景色を透明にするように変更。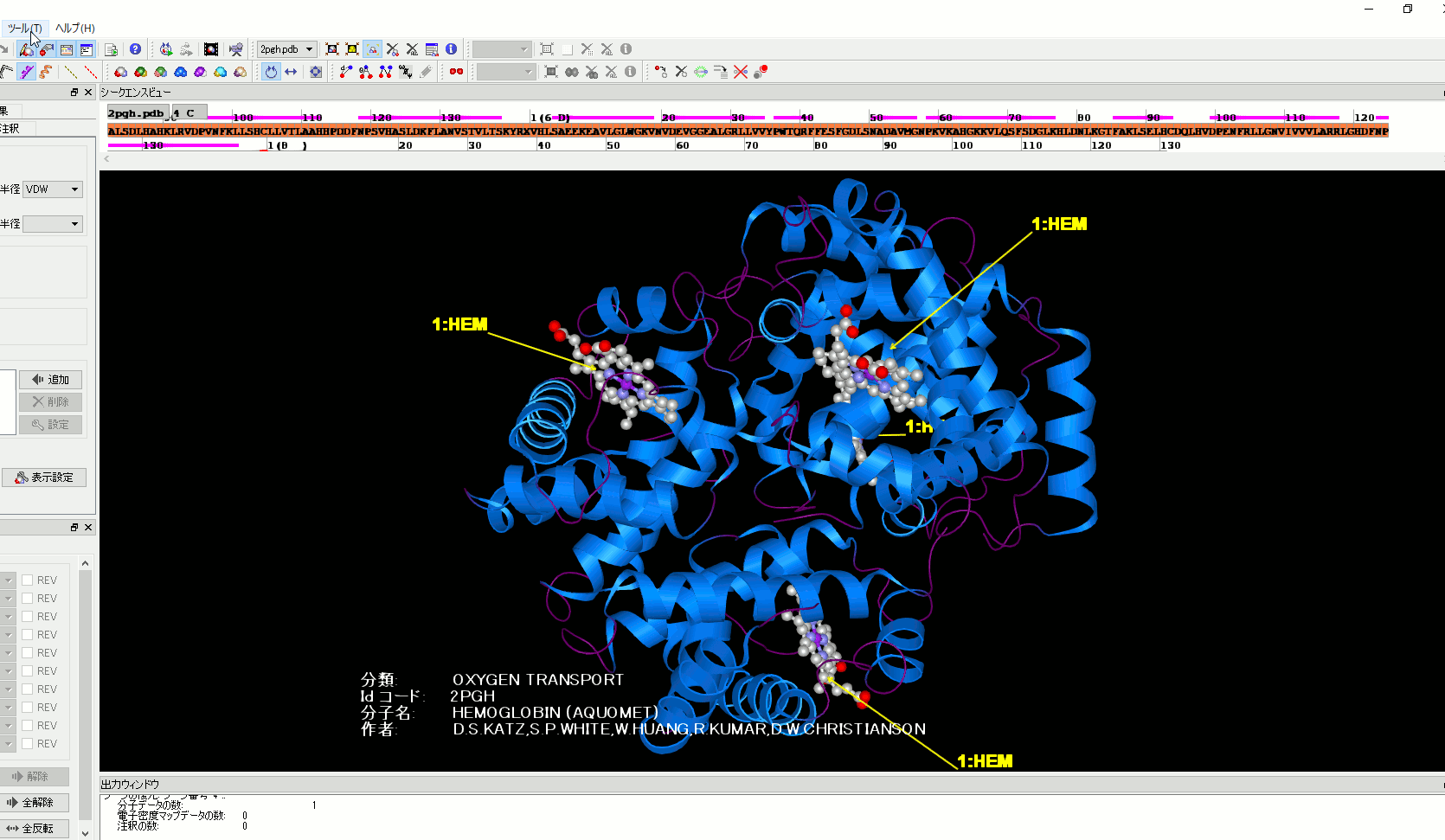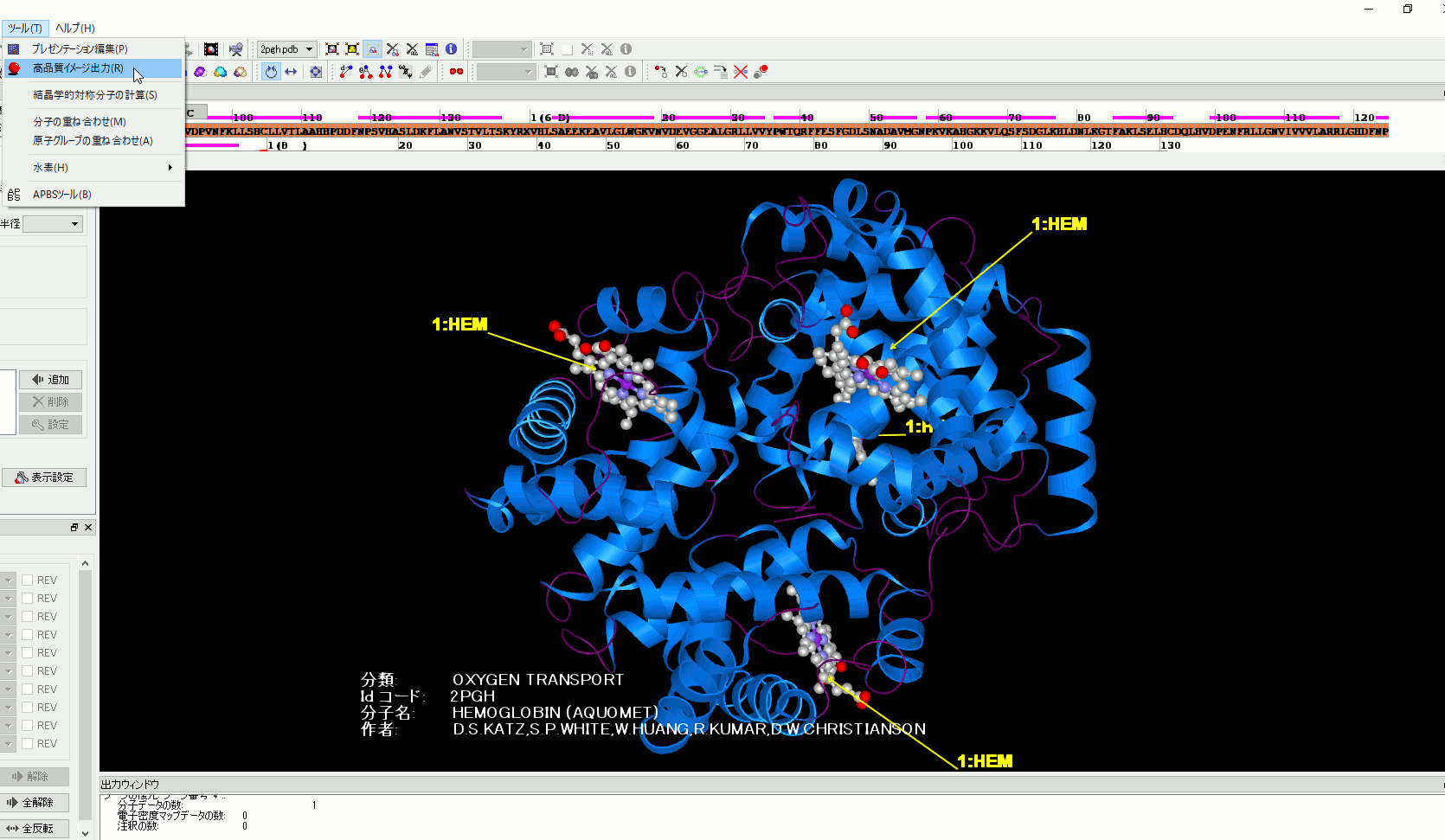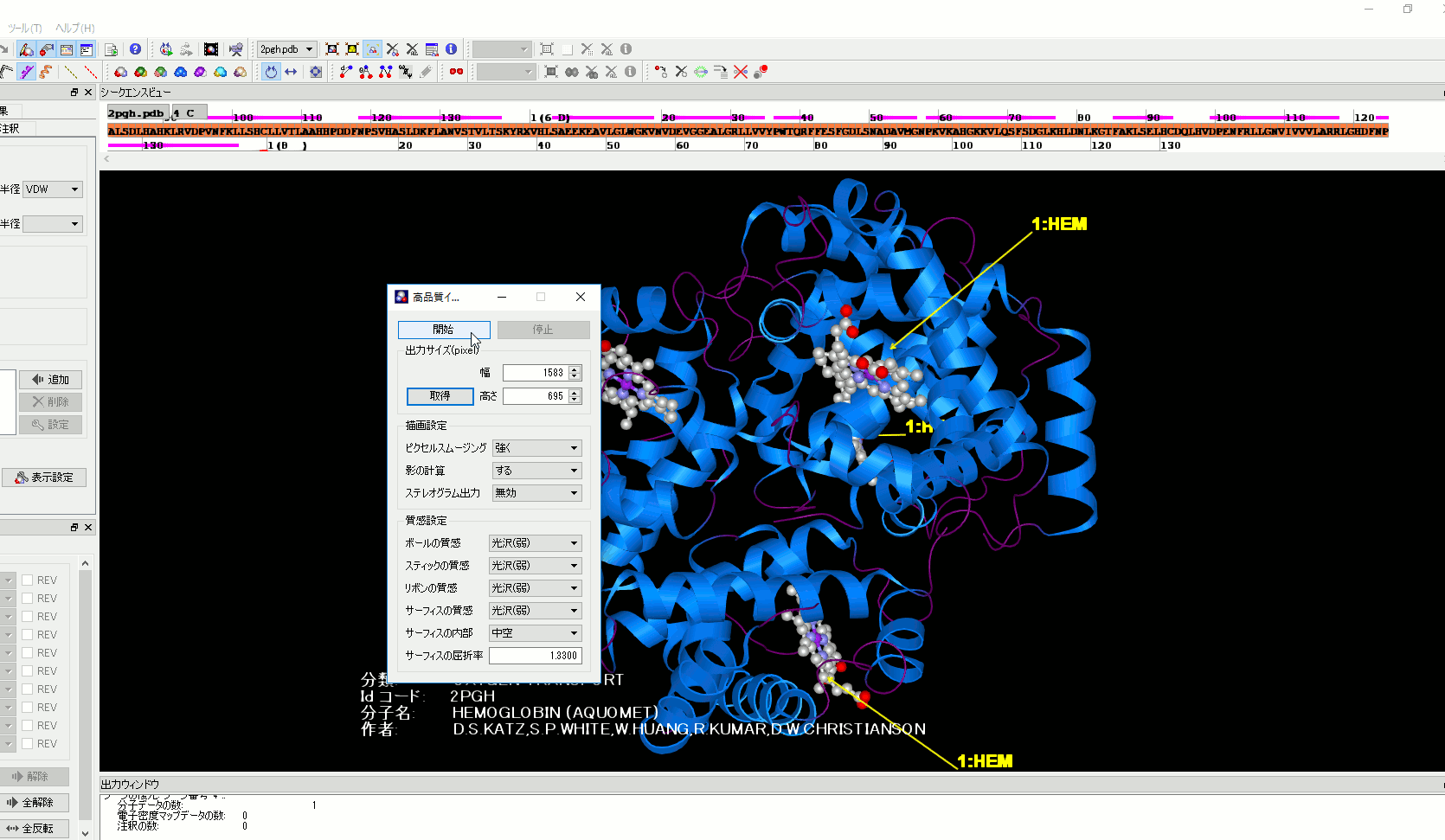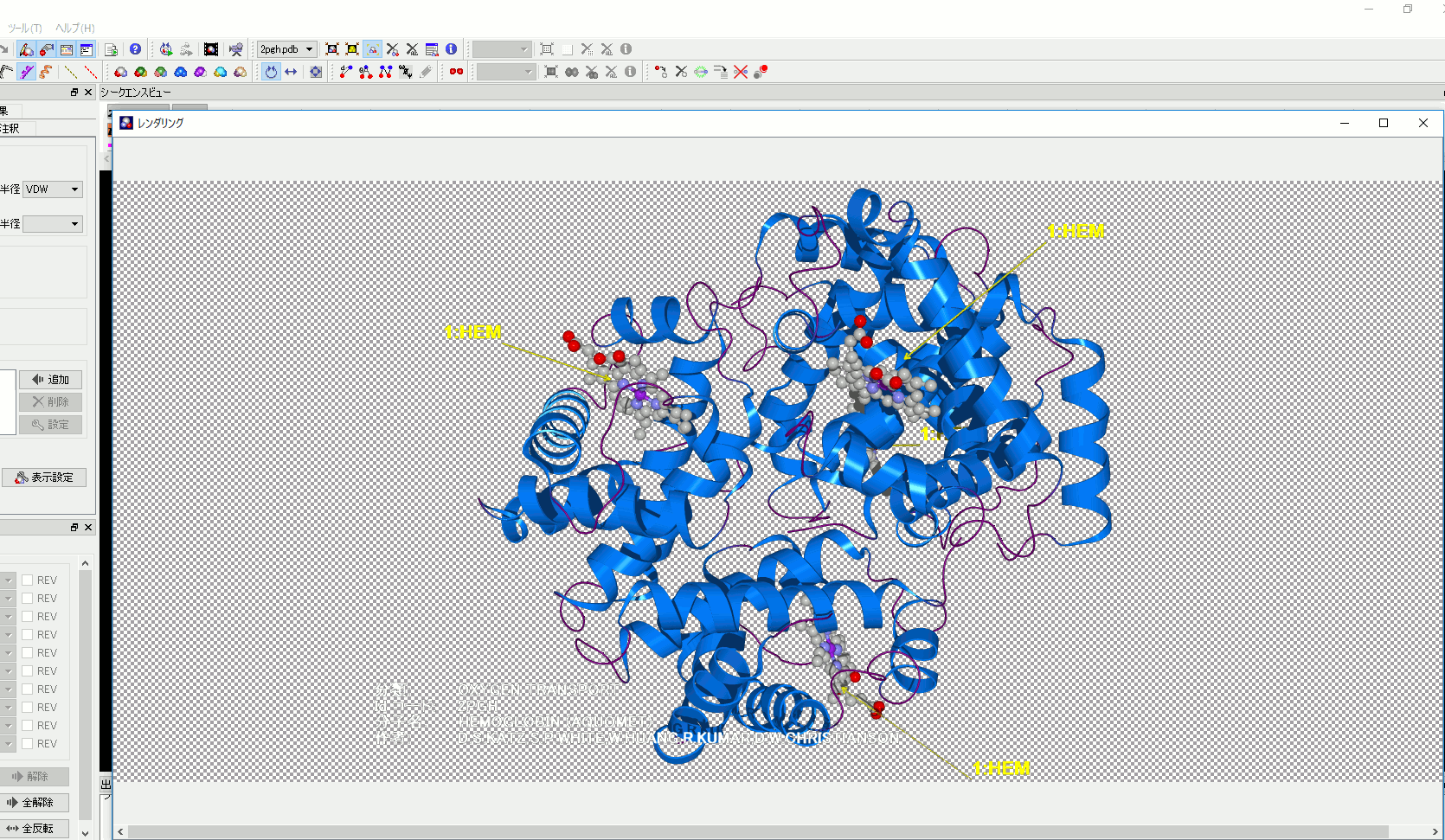
6. 分子の重ね合わせをアラインメントに沿って自動的に行うコマンドを追加。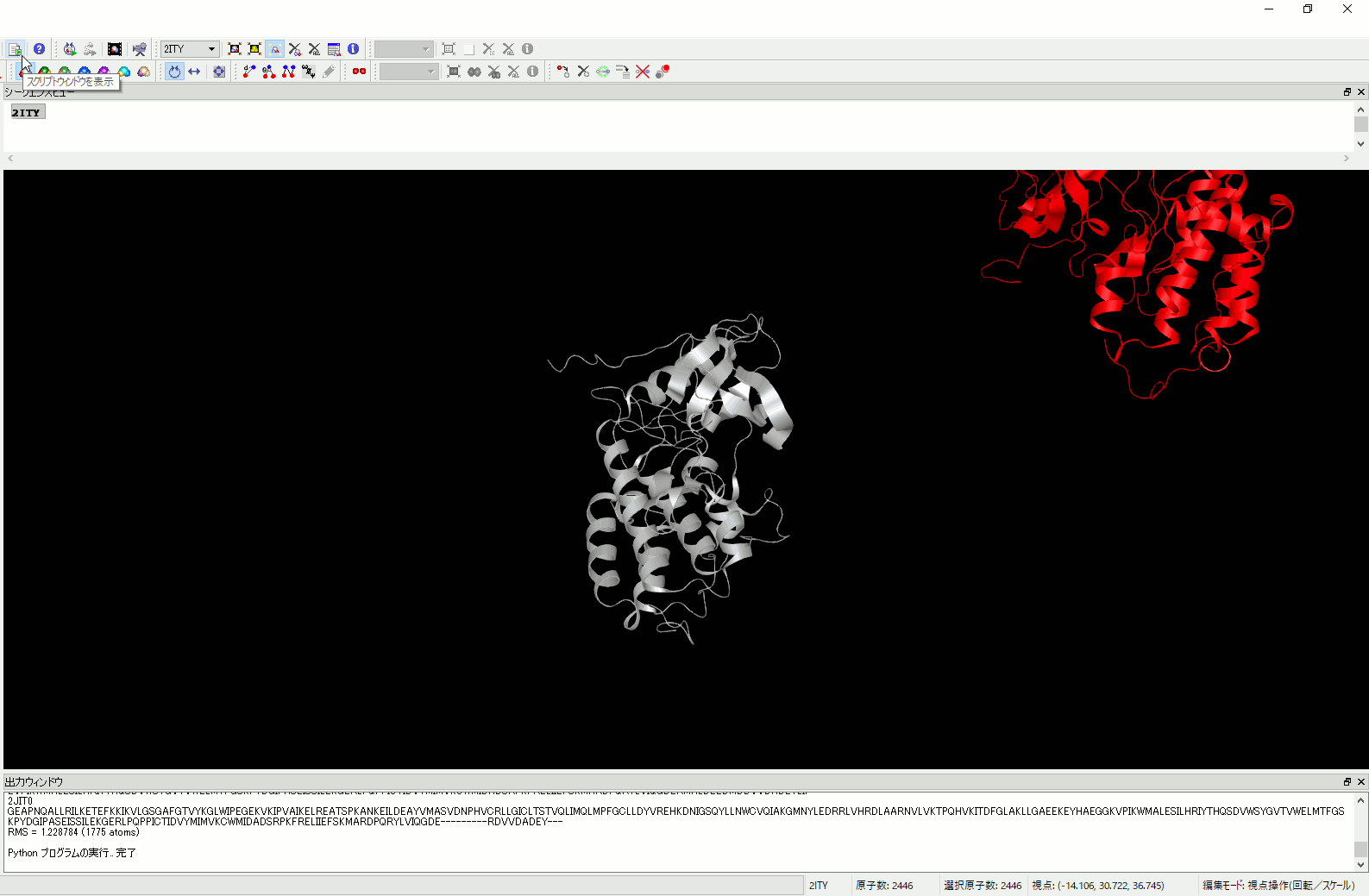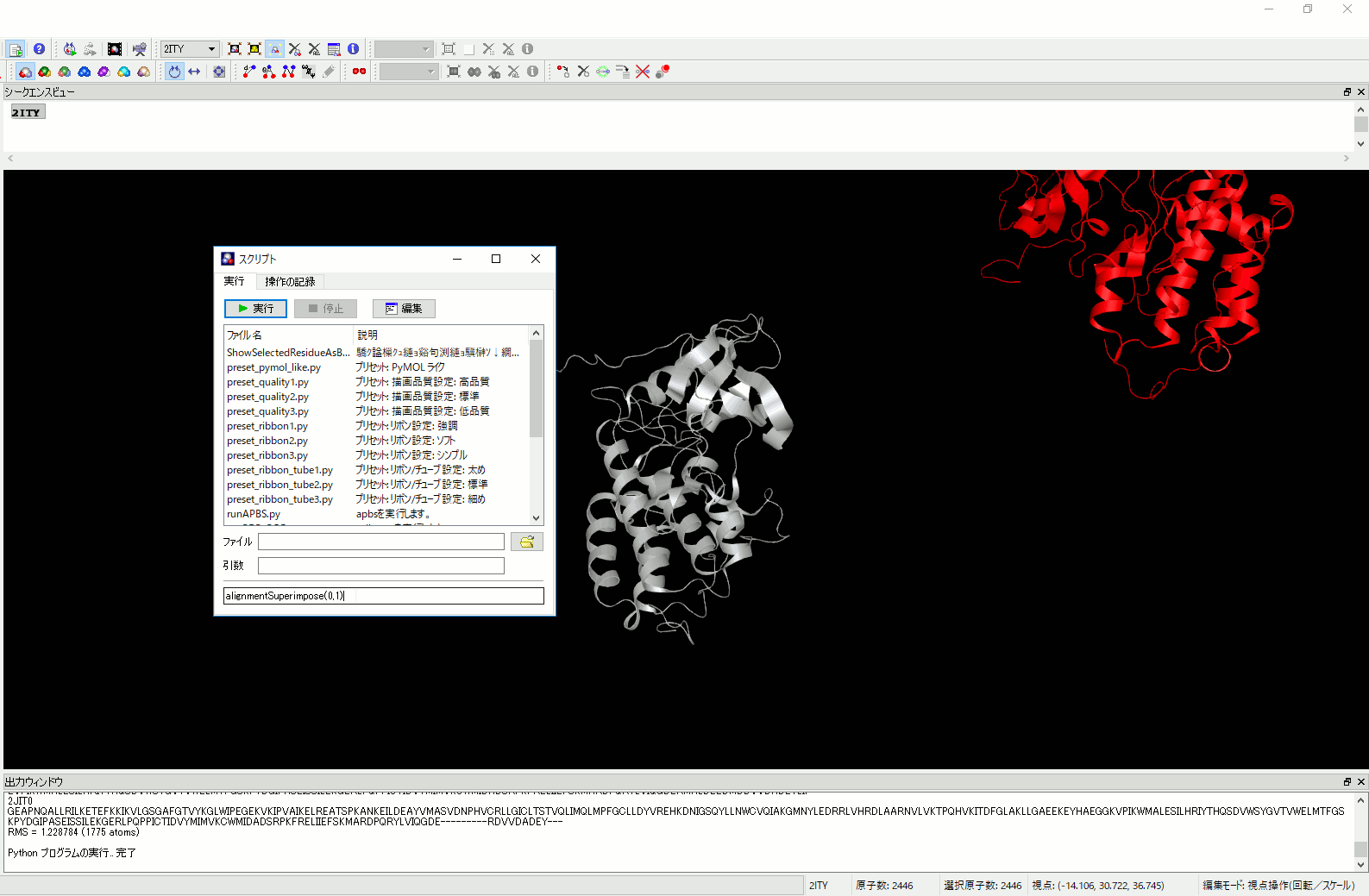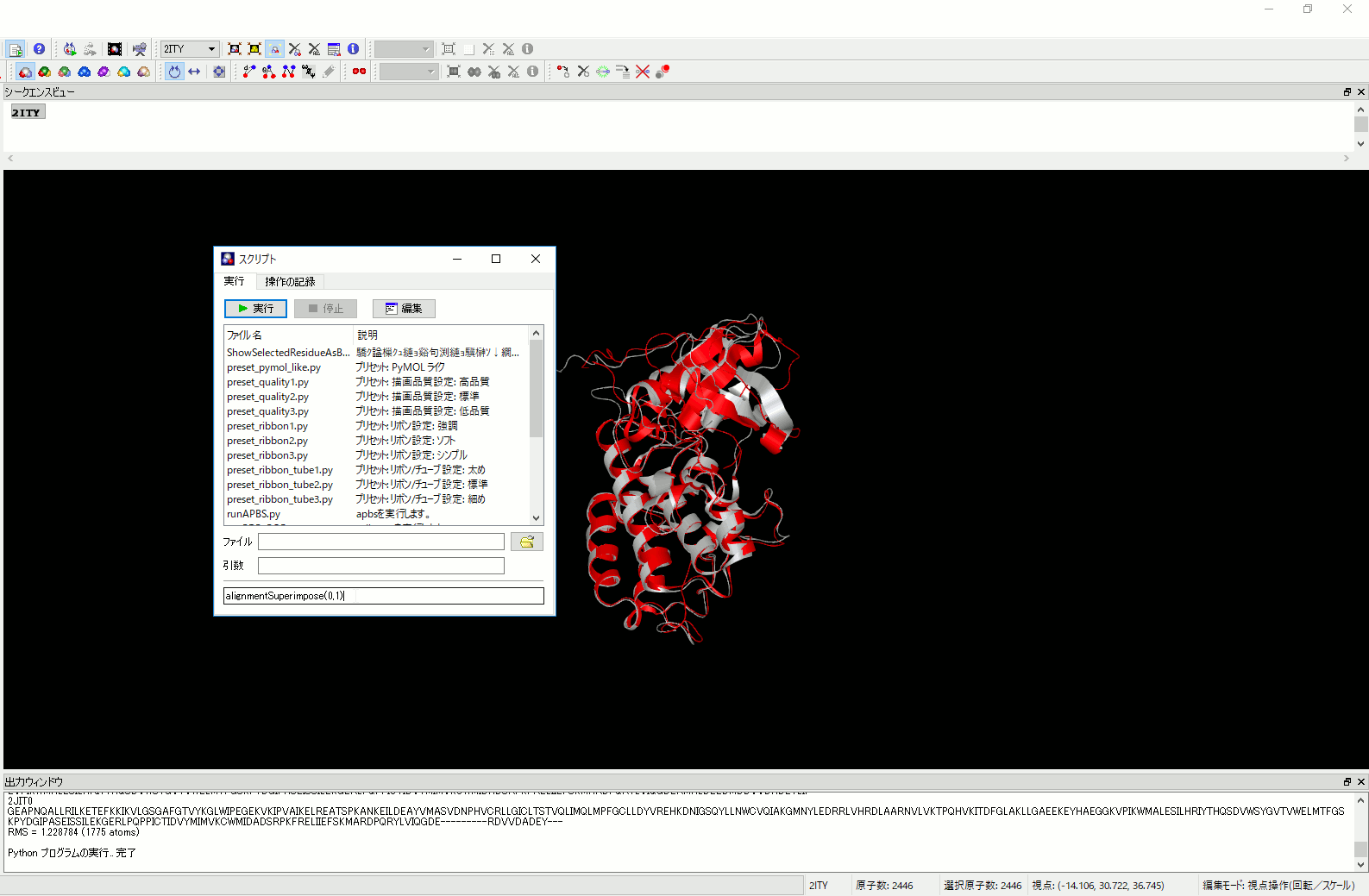
7. 結合の多重度を表示するとき芳香環を実線と破線で表示するように変更。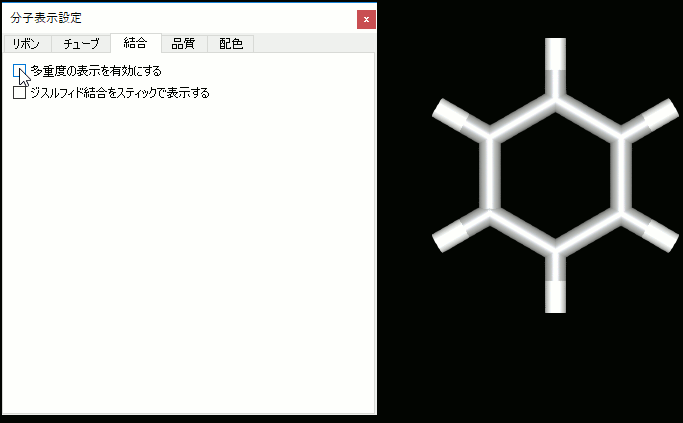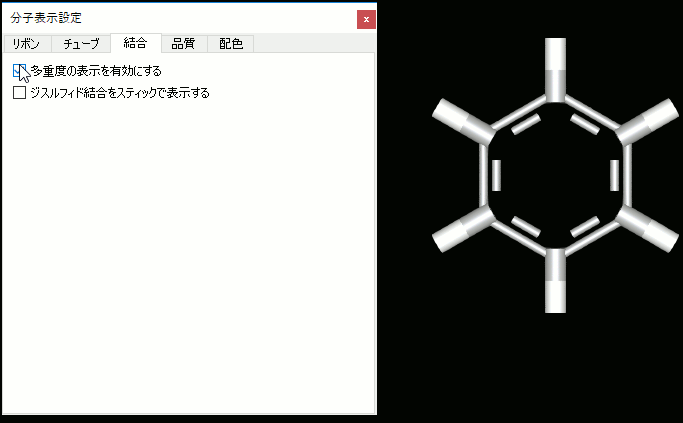
8. 低分子化合物の編集機能を追加。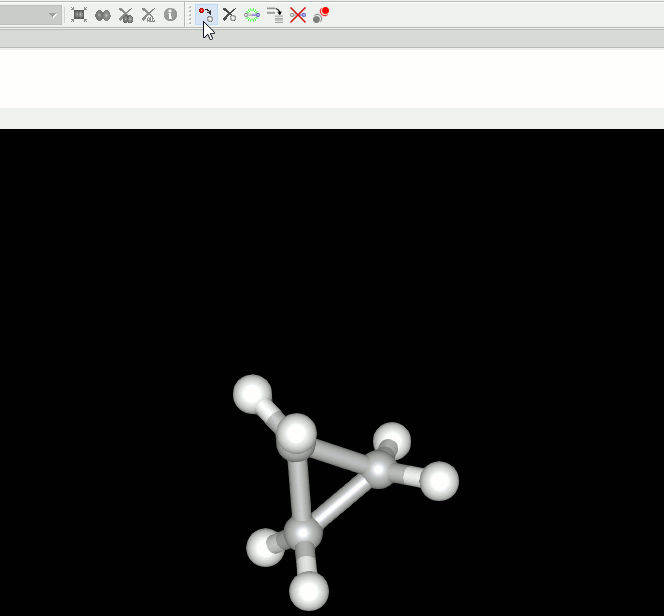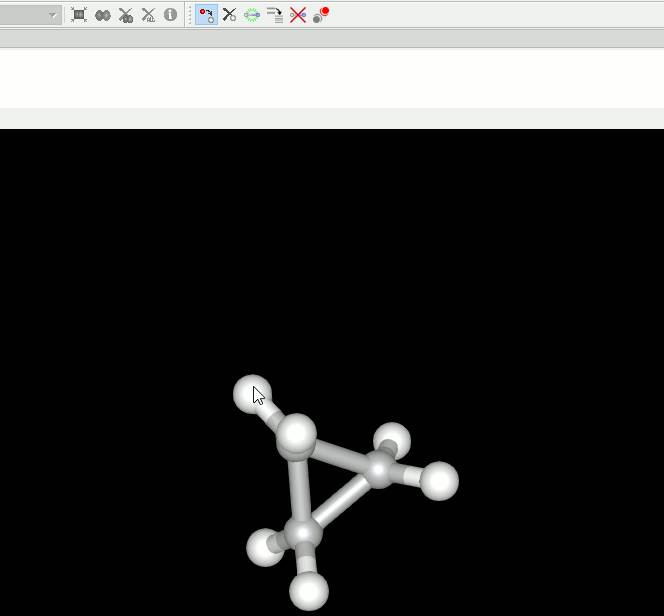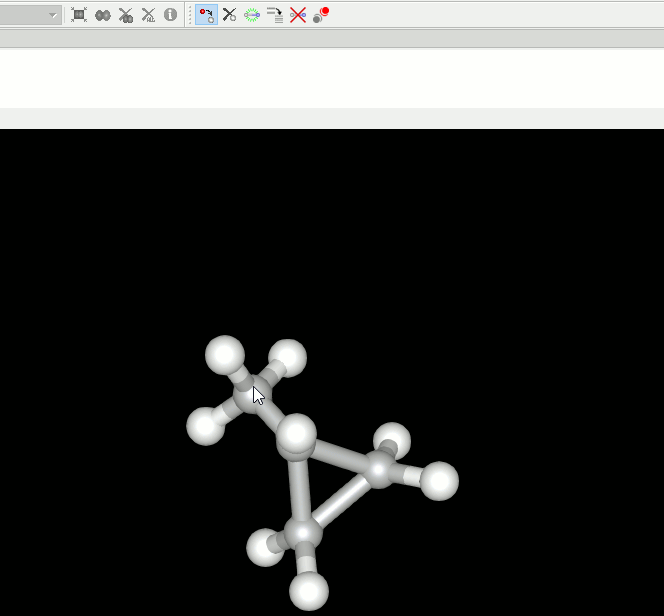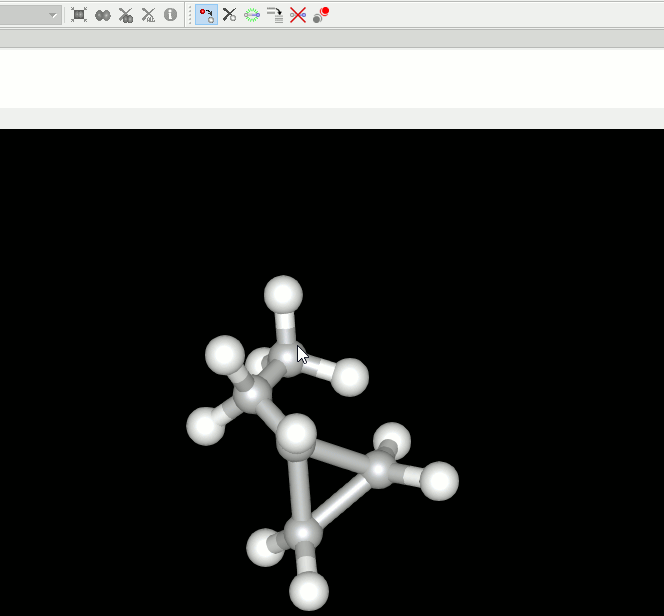
9. 分子をmol2形式で書き出す機能を追加。
MolFeat v5.1.1.25
新機能・修正点
MolFeatV5.1.1.25の新機能・修正点は以下のとおりです。
- シーケンスビューをフロート(メインのウィンドウから独立した状態)にした際に、Shiftキー/Ctrl(Mac版は⌘)キーによるシーケンスビューのアイテムの選択ができなくなる不具合を修正。
- チューブ表示の先端を半球状に変更。
- ボールをポイントスプライトで表示した時、色合いがおかしかったため修正。
- zSpaceで描画した際、左目右目が反転する不具合を修正。
- zSpaceで分子をピックした時にバイブレーションで知らせる機能を追加。
MolFeat v5.1 新機能・修正点
- フレームシーケンシャルの3D表示を、全画面で行える機能を追加。
- グラフィックスボードを利用しているマシンで、ポイントスプライトによる球の表示と、ポイントスプライトのON・OFFを切り替える機能を追加。
- 選択パネルの[種類]のタブに、水素・極性水素・非極性水素を選択する機能を追加。
- mol2/molファイルの読み込みで、ファイルに記載のない結合が追加される不具合を修正。
- pdb2pqr 2.0に対応。
- 分子の自動回転に首振り機能を追加。
- Retinaディスプレイで動画の作成や高品質イメージ出力以外の画像の書き出しを行うと左下4分の1だけが出力される不具合を修正。(Mac版の場合のみ)
- Qtライブラリの影響でドラッグ&ドロップしたファイルが読み込めなくなっていた不具合を修正。(Mac版 Yosemiteの場合のみ)
- その他細かい不具合を修正。
MolFeat v5.0
新機能・修正点
-
Gaussian CUBEファイルの読込機能を改良
- Gaussian CUBEファイルで読み込める原子の最大数を以前より多く設定
-
MolFeatコントロールの改良
- MolFeatコントロールで、Webに複数のコントロールを配置した場合、何も描画しなくなる不具合の修正
- mmCIF形式のデータの読込に対応
- Qtのライブラリを使用したGUIに移行
-
PDBファイルの保存機能の改良
- 原子数が99999以上の場合でも、PDBファイルを出力できるように修正
MolFeat v4.6
新機能・修正点
- 低分子の極性水素原子、非極性水素原子の選択
- GUI上からAPBSのグリッドの解像度を設定
- ムービー作成時のフレーム指定
- 静電ポテンシャルのカラーバーの表示/非表示
- 注釈の表示/非表示
- マウス操作によるZ軸回転
- スクリプトを使ったクリッピング幅の変更
- Cosgene のトラジェクトリの読込
- netcdfの読込
- PowerPoint の資料用の画像に上下または、左右にできるグレーの領域の透明化
MolFeat v4.5
新機能・修正点
- Gaussian CUBE ファイルの下記の表示に対応
- 分子軌道
- 静電ポテンシャル
- 電子密度
- PDB2PQR、APBS による静電ポテンシャル計算に対応
- ポアソンボルツマンソルバーによる静電ポテンシャル計算
- pH 値を考慮した計算
- 分子サーフィス形状上にポテンシャルを表示
- マルチグリッド形式をサポート
- OpenDX ファイル対応
- 任意平面上での静電ポテンシャルエネルギー分布の表示に対応
(APBS により静電ポテンシャル計算を実行した場合) -
シーケンスビューの改良
- 表示形式を切り替えたときに表示位置が保存されるような修正
MolFeat v4.0
新機能・修正点
-
トラジェクトリファイルローダー
- DCD 形式 (CHARMM、NAMD)
- TINKER形式
- GROMACS形式
-
選択機能の強化
以下のような選択式で原子選択ができるようになりました。- protein | protein aroundres:4.0 タンパクとその周囲4Å以内の残基
- $ & resname:ALA 現在選択中の原子で残基名が”ALA”の原子
-
スクリプト実行機能
Pythonスクリプトを使ってMolFeatを操作できるようになりました。ユーザーインターフェースからできる操作とほぼ同等の操作がスクリプトより可能です。 例えば以下のようなことができます。MolFeatコントロール上でもスクリプトを実行可能です。-
操作の自動化
データダウンロード → 原子選択 → 形状編集 → 高品質イメージ出力
といった一連の操作を複数の分子に対して自動的に実行 - 簡単な分子アニメーション
- 操作内容の記録機能
- ユーザーインターフェースに対しておこなった操作を記録し、その内容をPythonスクリプトとして出力できます。
-
操作の自動化
- 半透明ポリゴン描画処理の改良
-
シーケンスビューの機能強化
- シーンウィンドウ中でクリックした原子を、シーケンスビュー上にスクロールして表示します
- シーケンスビューの表示モードを切り替えたときに以前の表示位置を保ちます。
- 分子の分離機能の強化
1つの分子データを選択原子とそうでない原子で分離できます。 - 分子サーフィスへ独自色設定
スクリプトを介して、分子サーフィスへ頂点単位で独自色を設定できます。
MolFeat v3.6
新機能・修正点
- ムービーファイル作成機能
AVIファイルを作成します。 - 水素原子の付加と削除
- CHARMM PDB ファイル対応
原子数が99999個以上のPDBファイルを読み込めるようにしました。 - 選択原子から指定距離以内にある残基選択(原子選択)
- リボン/チューブの半透明表示
- 距離注釈の表示モード追加
単なる線分表示モードを追加 - 高品質イメージ出力機能の機能追加
MolFeat v3.5
新機能・修正点
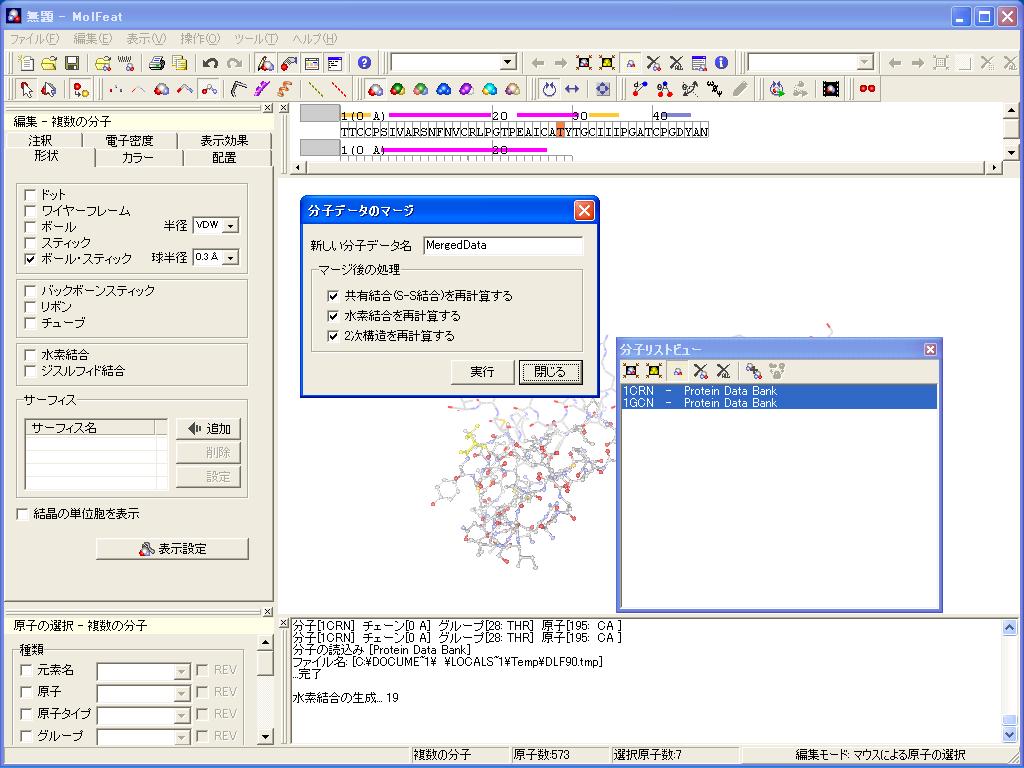
- 分子データのマージ
指定した複数の分子データをマージして1つの分子データにします。 - 分子データの分離
1つの分子データを分子鎖を最小単位として複数の分子データに分離します。 -
電子密度表示
読み込んだ1つの電子密度データに複数の表示領域を作成できるようになりました。 表示領域は、選択した複数の原子を中心に作成ができます。 また、これまでのワイヤーフレームの表示だけでなく、ポリゴン、ドット表示もできるようになりました。 - シーケンスビュー
シーケンスを折り返して表示するモードを追加しました。また、複数の分子データの表示にも対応しました。 - 疎水性インデックス値による色づけ
Kyte and Doolittle の疎水性インデックスでの色づけに対応しました。 - 主鎖2面角φ、ψのファイル出力
1つの分子データの2面角φ、ψをファイルに出力できるようになりました。gnuplot などでプロット可能な形式です。 - AMBER Restart ファイルへの対応
- 水素結合の破線ピッチの調整
- ジスルフィド結合のスティック表示対応
- 平行投影のサポート
- ステレオ表示モード
水平インターリーブ方式に対応しました。xpol などの表示装置で利用できる方式です - PDBv3.0への対応
MolFeat v3.0
新機能・修正点
- 新規ファイル作成、保存、undo、redo機能追加
- トラジェクトリファイルのアニメーション再生
- 指定した分子の対称構造の計算
PDBに記録された単位胞および空間群の情報を使って、指定した分子の対称構造を計算して表示します。 - 水素結合の自動計算強化
蛋白質内、核酸内だけでなく、すべての原子に対して水素結合を計算します。 - 選択機能の強化
指定原子を中心に半径何Å以内の原子選択をできるようにします。 - MolFeatコントロールの機能強化
自動回転の回転軸、回転スピードを指定できるようにします。 - シフトキーによる分子の平行移動機能追加
MolFeat v2.2
新機能・修正点
-
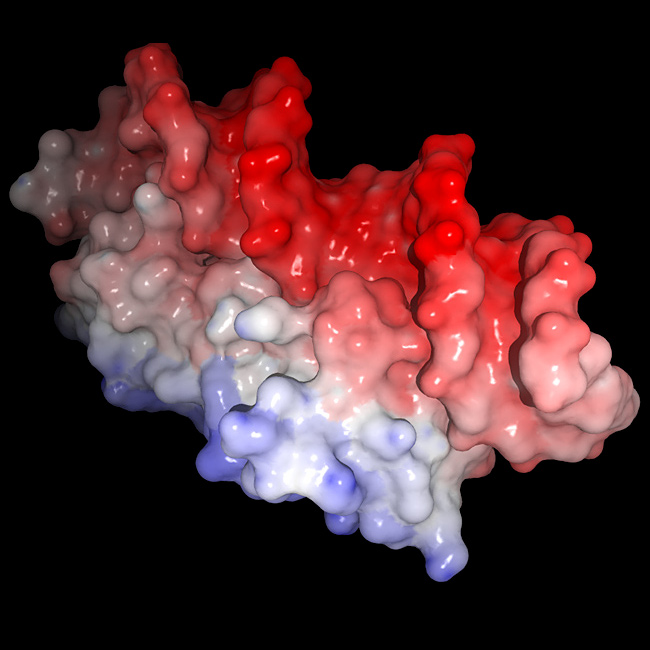
静電ポテンシャルの計算と可視化
タンパク質分子、核酸分子の静電ポテンシャルを計算し可視化します。 計算にはAMBERのテンプレートデータを使用しています。 配色設定により、静電ポテンシャル値に対応する色を編集可能です。 -
分子リストビュー
読み込んだ分子データを一覧で表示、選択するためのビューです。 このビューを使用すると編集対象分子を複数選択できるようになります。 - すべての分子形状のクリッピング
分子サーフィス形状以外に、ボール、リボンの形状などもクリップ可能です。 - 距離、角度、2面角計測結果の注釈生成
計測後、その結果を3次元空間中に注釈として残せます。 -
リボン、チューブ表示の機能強化
以下が強化されています。
- 幅、厚みの設定
- 表示品質の設定
- 表示のバリエーション追加
- 曲線補間処理の改善
-
ポイントミューテーション
シーケンスビューを使って、タンパク質分子、核酸分子内の残基を指定した 残基で置換え可能です。構造最適化などの処理はおこなっていません。
価格
| 定価 | USBライセンスキー 1ライセンス ¥98,000(税別) |
|---|
教育機関向けは3ライセンス付属します。
ひとつのサイト(同一敷地内)で複数のライセンスをご利用いただく場合、サイトライセンスの複数ユーザパッケージプランも準備しております。サイトライセンスをご希望の場合はこちらまでお問い合わせください。
ご注文方法
弊社は特定の代理店を設けておりません。基本的にお客様と直接取引をいたしております。ご注文の際は、こちらからお問い合わせください。
納品物について
MolFeatの納品物は、インストールメディア(CDROM) 1枚、USBライセンスキー 1個(アカデミックは3個) です。マニュアルはPDFファイルでご用意しており、必要に応じてお客様に印刷していただいております。